- Home
- Volume 43 (2025)
- Caractérisation génétique de deux espèces proches du sous-genre Clarias (Clarias) : C. gariepinus et C. anguillaris, à l’aide de marqueurs microsatellites au Burkina Faso
View(s): 141 (9 ULiège)
Download(s): 0 (0 ULiège)
Caractérisation génétique de deux espèces proches du sous-genre Clarias (Clarias) : C. gariepinus et C. anguillaris, à l’aide de marqueurs microsatellites au Burkina Faso

Résumé
Le développement de l’aquaculture africaine est en passe de connaitre un véritable succès grâce à l’utilisation de Clarias gariepinus (Burchell, 1822) dans certains pays subsahariens. Cependant, l’utilisation des individus sauvages dans les structures d’élevages pose problème à cause de sa forte ressemblance morphologique avec Clarias anguillaris (Linnée, 1758). Une étude menée au Burkina Faso a identifié ces deux espèces sur la base du nombre de branchiospines sans pour autant avoir la confirmation génétique de celles-ci. L’objectif de cette étude vise à répondre à deux questions fondamentales : (i) est-il possible de discriminer les deux espèces en utilisant les marqueurs microsatellites ? (ii) la variabilité du nombre de branchiospines est-elle influencée par les facteurs génétiques ou environnementaux ? Pour ce faire, 120 individus de C. anguillaris et C. gariepinus répartis dans 4 sites (Bama, Bala, Sourou et Kompienga) ont été examinés. Ainsi, sept marqueurs microsatellites (Cga01, Cga03, Cga05 Cga06, Cg07, Cga09 et Cga10) ont été utilisés pour caractériser la variabilité génétique de ces populations. Les résultats obtenus suite à une analyse de régression, montre une très forte corrélation entre le nombre de branchiospines et les cordonnées génétiques des individus obtenus à la suite d’une Analyse Factorielle de Correspondance (AFC). Cependant, l’analyse de la différentiation génétique à partir des statistiques de Wright montre une très faible différentiation entre les deux espèces avec un FST moyen estimé à 0,042 (P = 0,02). L’utilisation d’autres techniques, telles que l’usage de l’ADN mitochondrial devrait permettre une discrimination nette de ces deux espèces.
Abstract
The development of African aquaculture is becoming a real success thanks to the use of Clarias gariepinus (Burchell, 1822) in some sub-Saharan countries. However, the use of wild individuals in breeding structures is problematic because of its morphological resemblance to Clarias anguillaris (Linnée, 1758). A study in Burkina Faso identified these two species on the basis of the number of gill rakers without having the genetic confirmation of these. The purpose of this study is to answer two fundamental questions: (i) is it possible to discriminate between the two species using microsatellite markers? (ii) is the variability in the number of gill rakers influenced by genetic or environmental factors? To do this, 120 individuals of C. anguillaris and C. gariepinus distributed in 4 sites (Bama, Bala, Sourou and Kompienga) were examined. Thus, seven microsatellite markers (Cga01, Cga03, Cga05 Cga06, Cg07, Cga09 and Cga10) were used to characterize the genetic variability of these populations. The results obtained following a regression analysis, shows a very strong correlation between the number of gill rakers and the genetic coordinates of individuals obtained following a Correspondence Factor Analysis (CFA). However, the analysis of genetic differentiation from Wright's statistics shows a very low differentiation between the two species with an estimated mean FST of 0.042 (P = 0.02). Indeed, the use of other techniques, such as the use of mitochondrial DNA should allow a clear discrimination of these two species.
Table of content
Introduction
1L’élevage des espèces du sous-genre Clarias (Clarias) et en particulier Clarias gariepinus (Burchell, 1822) préoccupe depuis quelques années les scientifiques et les responsables du développement de certains pays africains et dans le monde (Rukera Tabaro et al., 2005; Hecht, 2013). Ainsi de nombreuses études ont été entreprises et l’état actuel des connaissances sur la caractérisation morphologique de ces espèces est relativement avancé (Teugels, 1982; Teugels, 1986; De Graaf et Janssen, 1996; Paugy et al., 2003; Compaoré et al., 2015). Tous ces travaux ont reconnu que parmi les variables morphologiques ayant servi à leur identification, seul le nombre de branchiospines sur le premier arc branchial en relation avec la longueur standard permet de les discriminer avec succès. D’autres critères tels que la morphologie de la fontanelle frontale (Levèque et al, 1992) et la morphologie des cleithra (Benech et Teugels, 1997) ont été utilisés pour leur identification mais reste minime et très peu utilisable sur le terrain. Cependant, l’utilisation des caractères morphologiques pour l’identification des espèces présentent des limites car ils sont pour la plupart sensibles aux conditions environnementales (Marchal, 1964; Boisneau, 1990). Chez les deux espèces, et surtout chez C. gariepinus, ce nombre est lié à la taille; mais aussi, à une variation clinale de ce caractère comme rapporté par Teugels (1982). Si le nombre de branchiospines peut être déterminé par une variation environnementale, la différenciation à partir de ce seul critère est peu solide (Rognon et al., 1998). Ainsi, une discrimination fiable nécessiterait de nouvelles approches utilisant des marqueurs neutres non influencés par les conditions environnementales. C’est en ce sens que Teugels et al. (1992) ont utilisé l’approche protéique en examinant la variation électrophorétique de 13 loci de protéines dans deux populations d'Afrique de l'Ouest des deux espèces. Agnèse et al. (1997) ont utilisé des marqueurs iso enzymatiques et microsatellite pour différencier les deux espèces. Tous ces résultats ont montré que les populations sympatriques étaient différentes de manière significative et que les groupements morphologique et génétique étaient congruents.
2En revanche, si les études de Rognon et al. (1998) confirment les différences morphologiques qui existent entre les deux espèces, elles émettent néanmoins des réserves sur la traduction de la variabilité du nombre de branchiospine en différenciation génétique. D’autres travaux, utilisant divers marqueurs de l’ADN mitochondrial comme le cytochrome b (cyt b), le cytochrome oxydase c sous-unité I (COI), le ND5/6 ainsi que l'ARNr 16S pour étudier les relations phylogénétiques de plusieurs espèces de la famille des Clariidae ne mentionnent que Clarias gariepinus (Devaere et al., 2007; Mohindra et al., 2007; Wong et al., 2011; Yu et Quilang, 2014).
3Tout récemment, des travaux portant exclusivement sur les caractères morphologiques ont révélé l’existence des deux espèces du sous-genre Clarias (Clarias) au Burkina Faso (Compaoré et al., 2015). Au regard des controverses ci-dessus sur l’identification précise de ces deux espèces du point de vue génétique, il apparait nécessaire de procéder à leur caractérisation génétique avec des marqueurs neutres vis-à-vis de la sélection naturelle. Ceci permettra de savoir quelle espèce est utilisée dans les structures d’élevage aquacole surtout en Afrique où la confusion existe. De ce fait, les marqueurs microsatellites se sont révélés comme des marqueurs de choix pour les études d’identification des espèces, des tests de paternités et de structuration des populations (Ellegren, 2004). En outre, ils se sont avérés être des outils précieux pour la caractérisation et l'évaluation de la diversité génétique en raison de leur caractère très polymorphe, la facilité et la rapidité des protocoles de révélation. Plusieurs travaux ont utilisé ces marqueurs pour identifier des espèces très apparentées et sympatriques (Fritzner et al., 2001; Bonhomme et al., 2002; Renshaw et al., 2006). C’est ce qui justifie leur choix dans le présent travail pour identifier les deux espèces du sous-genre Clarias-clarias.
4La présente étude, à travers l'analyse de la variabilité de 7 marqueurs microsatellites, vise à répondre à deux questions fondamentales : (i) est-il possible de discriminer les deux espèces en utilisant les marqueurs microsatellites ? (ii) la variabilité du nombre de branchiospines est-elle influencée par les facteurs génétiques ou environnementaux ?
Matériel et Méthodes
Matériel
Matériel biologique
5Le matériel biologique a concerné 330 individus de Clarias sp ayant fait au préalable l’objet d’une étude d’identification morphologique (Compaoré et al., 2015). Il ressort de cette étude que 275 et 55 échantillons ont été identifiés comme Clarias anguillaris et Clarias gariepinus respectivement sur la base de leurs nombres de branchiospines. D’autres critères ayant fait l’objet d’identification des deux espèces sont résumés dans le tableau 1. Ces échantillons proviennent des plans d’eau issus des trois bassins versants qui constituent le réseau hydrographique du Burkina Faso (figure 1). Le tableau 2 résume la provenance et l’effectif de chaque espèce en fonction du site d’étude et de son bassin versant. Sur chaque échantillon, un morceau d’environ 1 cm2 de la nageoire caudale a été prélevé et conservé dans des tubes Eppendorf de 1,5 ml contenant de l’éthanol absolu jusqu’à utilisation.
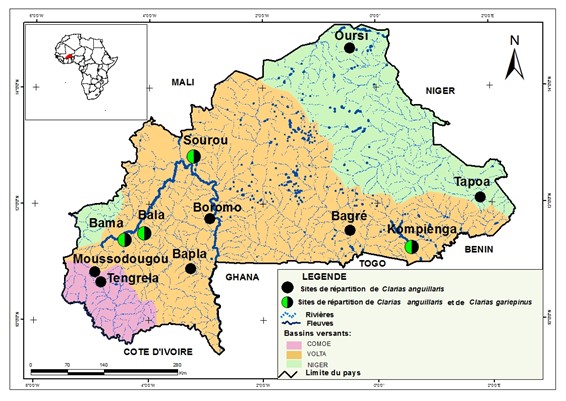
Figure 1 : Sites de répartition de Clarias gariepinus et Clarias anguillaris suivant les trois bassins versants du Burkina Faso (Source : BNDT 2002, IGB/Enquêtes_Terrain).
6-
Tableau 1 : Résumé des caractères distinctifs entre C. gariepinus et C. anguillaris
|
Description |
C. gariepinus |
C. anguillaris |
Auteurs |
|
Nombre de branchiospines |
28-120 |
13-39 |
Compaoré et al., (2015) |
|
24 -110 |
16-50 |
Lévêque et al., (1992) |
|
|
Fontanelle frontale |
longue et étroite |
courte et large |
Lévêque et al., (1992) |
|
Cleithrum lisse |
0,67 |
0,22 |
Benech et Teugels (1993) |
|
Cleithrum ponctué |
0 |
11% |
|
|
Cleithrum strie |
0,33 |
0,24 |
|
|
Cleithrum ponctué et strié |
0 |
0,43 |
|
|
Cleithrum étroit |
0,67 |
0,14 |
|
|
Cleithrum intermédiaire |
0,13 |
0,14 |
|
|
Cleithrum large |
0,2 |
0,72 |
Méthodes
Extraction de l’ADN génomique
7L’extraction de l’ADN total a été faite à partir d’un échantillon de 1 cm² environ de tissus de la nageoire caudale avec le Kit de purification Promega®. Ces tissus ont été digérés dans un mélange composé de 200 µl de solution de lyse (Nuclei Lysis), de 20 µl de solution de protéinase K et de 420 µl de solution d’EDTA (Ethylène Diamine Tétra Acétique). Le mélange, une fois agité, a été passé au vortex puis mis en incubation pendant une nuit (18 heures du soir à 07 heures du matin) à 56°C dans une étuve. Après une digestion complète des tissus, un ajout de 200 µl de solution de protéine de précipitation a été effectué suivi d’une centrifugation à 14 000 rpm pendant 4 mn. Ensuite, la phase aqueuse a été récupérée et transférée dans des tubes contenant 600 µl d’isopropanol par précipitation. L’ADN se dépose sous forme de culot après une seconde centrifugation à 14 000 rpm pendant 4 mn. L’ADN ainsi obtenu a été lavé dans de l’éthanol à 70% puis réhydraté dans 150 µl de solution de Tris-EDTA. L’ADN est ensuite portée à 65°C pendant une heure et puis conservée à -20°C jusqu’au moment de son utilisation.
Tableau 2 : Nombre d’individus de Clarias anguillaris et Clarias gariepinus collectés par site d’étude.
|
Bassin versant |
Sites d’étude |
Total des 2 espèces collectées |
Nombre de Clarias anguillaris collecté |
Nombre de Clarias gariepinus collecté |
|
Comoé |
Tengrela |
30 |
30 |
0 |
|
Moussodougou |
30 |
30 |
0 |
|
|
Volta |
Bama |
30 |
7 |
23 |
|
Bala |
30 |
26 |
4 |
|
|
Bapla |
30 |
30 |
0 |
|
|
Sourou |
30 |
23 |
7 |
|
|
Boromo |
30 |
30 |
0 |
|
|
Bagré |
30 |
30 |
0 |
|
|
Kompienga |
30 |
9 |
21 |
|
|
Niger |
Tapoa |
30 |
30 |
0 |
|
Oursi |
30 |
30 |
0 |
|
|
Total |
330 |
275 |
55 |
Choix des marqueurs microsatellites
8Dans le cadre de ce travail, dix marqueurs microsatellites ont été sélectionnés pour identifier et caractériser la variabilité génétique des deux espèces du sous-genre Clarias (Clarias). Ces marqueurs ont été choisis dans le site de GenBank http://www.ncbi.nlm.nih.gov/genbank/. Ils ont été isolés pour caractériser Clarias gariepinus (Galbusera et al., 1996). Après les tests d’essais (variation des températures, durée d’hybridation et des produits d’amplification) sept marqueurs ont été retenus sur la base qu’un marqueur permet de visualiser au moins 50% des profils après migration des produits PCR sur le séquenceur Licors. Ce sont : Cga01, Cga03, Cga05 Cga06, Cg07, Cga09 et Cga10. Les numéros de ces marqueurs microsatellites dans Genbank et leurs caractéristiques sont consignés dans le tableau 3.
Amplification des microsatellites par PCR
9Les réactions de PCR ont été effectuées avec un thermocycleur Biometra. Le programme d’amplification consiste en une dénaturation pendant 3 min à 94°C, suivie de 35 cycles successifs. Chaque cycle comprend une succession de trois phases : une dénaturation à 94 °C pendant 30 s, une phase d’hybridation à la température optimale déterminée entre 59-65 °C pendant 30 s selon l’amorce et une élongation à 72°C pendant 45 s. Enfin, une dernière étape d’élongation à 72°C pendant 2 min et une phase de refroidissement à 4 °C. Le volume réactionnel utilisé pour chaque échantillon de 13,6 µl contient : 6,1µl d’eau distillée (H20); 1,25 µl de Tampon 10X; 1 µl de dNTP (2,5 mM); 2,5 µl de MgCL2 (25 mM); 0,25µl de AMF_M13 (10 µM); 0,25 µl de AMR (10 µM); 0,5 µl de Taq (5u/µl); 0,15 µl de Dye 700 (10 µM) et 2 µl d’ADN génomique. Les produits PCR amplifiés ont été dénaturés sur une plaque chauffante à 94°C pendant 3 minutes et déposés sur un gel d’acrylamide à 6,5% préalablement monté sur un séquenceur Li-Cor. Les allèles sont ensuite séparés par électrophorèse verticale haut voltage (1500 volts pendant 1 heure 30 minutes) en fonction de leur taille. La détection des allèles a été faite grâce aux deux lasers du séquenceur Li-Cor et la visualisation de la migration a été possible sur un ordinateur connecté au séquenceur.
Tableau 3 : Caractéristiques des marqueurs microsatellites utilisés sur les échantillons de Clarias gariepinus. (Source : Galbusera et al., 1996)
|
Locus |
Amorces (5’–3’) |
Motif (5’-3’) |
Température d'hybridation (°C) |
N° Genebank |
|
Cga01 |
Cga01-F : GGCTAAAAGAACCCTGTCTG Cga01-R: TACAGCGTCGATAAGCCAGG |
(GT)15 |
59 |
U30862 |
|
Cga03 |
Cga03-F: CACTTCTTACATTTGTGCCC Cga03-R : ACCTGTATTGATTTCTTGCC |
(AC)21 |
56 |
U30864 |
|
Cga05 |
Cga05-F : TCCACATTAAGGACAACCACCG Cga05-R : TTTGCAGTTCACGACTGCCG |
(CA)11N2(CA)2 |
60 |
U30866 |
|
Cga06 |
Cga06-F : CAGCTCGTGTTTAATTTGGC Cga06-R : TTGTACGAGAACCGTCCAGG |
(AC)4N2(AC)9N6(AC)6 |
60 |
U30867 |
|
Cga07 |
Cga07-F : TTGTGTAACTCACATTACCGG Cga07-R : GTTTAGAAAATGTTTACGCCG |
(AC)13 |
62 |
U30868 |
|
Cga09 |
Cga09-F : CGTCCACTTCCCCTAGAGCG Cga09-R : CCAGCTGCATTACCATACATGG |
(CA)2N2(CA)15 |
59 |
U30870 |
|
Cga10 |
Cga10-F : GCTGTAGCAAAAATGCAGATGC Cga10-R : TCTCCAGAGATCTAGGCTGTCC |
(GT)11N1(GT)6N2(GT)4 |
65 |
U30871 |
Analyse des données
10De nombreuses données manquantes ont été observées, combinées à des fort déficits en hétérozygotes, tels que mesurés par le FIS de Wright (1965) dans chaque espèce et pour chaque site. Dans un premier temps, un test a été fait pour savoir si les allèles nuls sont susceptibles d'expliquer ces déficits et données manquantes. Le logiciel Micro-checker (Van Oosterhout et al., 2004) a été utilisé pour estimer la présence d’allèles nuls qui pourrait influencer les écarts panmictiques de Hardy-Weinberg. La méthode consiste à estimer la fréquence d’allèles nuls susceptible d’expliquer, dans chaque sous-échantillon et pour chaque locus, les déficits en hétérozygotes observés. L’indépendance statistique des loci a été testé par la méthode des déséquilibres de liaison entre loci pour chaque espèce par site et dans l’ensemble à l’aide du logiciel Fstat (Goudet, 2003). La différenciation génétique a été testée entre les deux espèces dans chacun des sites où elles sont en sympatrie. Pour ce faire, le FST de Wright (1965), qui mesure le degré de subdivision entre sous-échantillons a été estimé. Ceci a été fait avec l'estimateur non biaisé θ de Weir et Cockerham (Weir et Cockerham, 1984).
11L'effet Wahlund correspond à un déficit d'hétérozygotes comparé aux attendus du modèle de Hardy-Weinberg, provenant de la présence, dans un même échantillon, d'individus appartenant à des populations génétiquement différenciées. La méthode la plus standard d'estimer cette déviation est le calcul du FIS de Wright (1965) qui mesure la consanguinité relative des individus par rapport à la consanguinité des sous-échantillons. Le FIS moyen des deux espèces a été estimé séparément et en les confondant (poolant) dans chaque site à l'aide de l'estimateur f de Weir et Cockerham (1984). Le FIS_séparées et le FIS_poolées a été comparé en procédant par un test de Wilcoxon pour données appariées (appariement par le locus et le sous-échantillon).
12Afin de vérifier la pertinence du nombre de branchiospines comme marqueur de différenciation entre les deux espèces, la corrélation entre la distance génétique entre individus et la différence de nombre de branchiospines a été mesurée, en corrigeant l'effet géographique. Pour vérifier si le nombre de branchiospines n'est pas expliqué par les facteurs environnementaux, un model linéaire généralisé (GLM) a été généré sous R avec la formule suivante: NombreBranchiospine~Longitude+Latitude+Bassin avec une erreur log normale (modèle de Poisson). Le modèle a été simplifié par une analyse de variance (Anova 1) suivie d’un test de Chi-2 appliquée au modèle pour déterminer la probabilité avec laquelle chaque facteur explique le nombre de branchiospines. Enfin, le logiciel MEGA 5.05 (Tamura et al., 2011) a été utilisé pour générer un arbre phylogénétique à partir de la distance de corde de Cavalli-Sforza et Edwards (1997).
Résultats
Estimation des fréquences d’allèles nuls avec Micro-checker
13Les tableaux 4, 5, et 6 présentent les résultats des fréquences d’allèles nuls respectivement pour Clarias anguillaris, Clarias gariepinus et les deux espèces regroupées.
14L’analyse de ces tableaux montre que tous les loci présentent des P-values, non significatives. Ce qui signifie que les allèles nuls sont suffisants pour expliquer les déficits en hétérozygotes observés au niveau de ces loci.
Tableau 4 : Synthèse des résultats de Micro-checker des 7 loci chez Clarias anguillaris.
|
Locus |
PB2² |
N |
N' |
Blancs observés |
Blancs attendus |
p-value |
|
Cga01 |
0,05 |
42 |
55 |
13 |
2,52 |
1 |
|
Cga03 |
0,05 |
42 |
55 |
13 |
2,98 |
1 |
|
Cga05 |
0,1 |
42 |
55 |
13 |
5,31 |
0,99 |
|
Cga06 |
0,41 |
24 |
55 |
31 |
22,39 |
0,99 |
|
Cga07 |
0,03 |
47 |
55 |
8 |
1,44 |
1 |
|
Cga09 |
0,16 |
35 |
55 |
20 |
8,51 |
1 |
|
Cga10 |
0,05 |
46 |
55 |
9 |
2,95 |
0,99 |
15-
Tableau 5 : Synthèse des résultats de Micro-checker des 7 loci chez Clarias gariepinus.
|
Locus |
PB2² |
N |
N' |
Blancs observés |
Blancs attendus |
p-value |
|
Cga01 |
0,42 |
28 |
51 |
23 |
21,62 |
0,71 |
|
Cga03 |
0,27 |
38 |
51 |
13 |
13,88 |
0,46 |
|
Cga05 |
0,54 |
29 |
51 |
22 |
27,43 |
0,08 |
|
Cga06 |
0,23 |
43 |
51 |
8 |
11,71 |
0,14 |
|
Cga07 |
0,07 |
47 |
51 |
4 |
3,53 |
0,72 |
|
Cga09 |
0,1 |
45 |
51 |
6 |
5,07 |
0,76 |
|
Cga10 |
0,19 |
40 |
51 |
11 |
9,55 |
0,76 |
16-
Tableau 6 : Synthèse des résultats de Micro-checker des 7 loci chez les deux espèces confondues de Clarias
|
Locus |
PB2² |
N |
N' |
Blancs observés |
Blancs attendus |
P-value |
|
Cga01 |
0,33 |
77 |
120 |
43 |
39,89 |
0,76 |
|
Cga03 |
0,24 |
87 |
120 |
33 |
28,95 |
0,83 |
|
Cga05 |
0,41 |
78 |
120 |
42 |
49,57 |
0,09 |
|
Cga06 |
0,46 |
74 |
120 |
46 |
55,09 |
0,06 |
|
Cga07 |
0,12 |
104 |
120 |
16 |
14,04 |
0,76 |
|
Cga09 |
0,24 |
88 |
120 |
32 |
28,72 |
0,79 |
|
Cga10 |
0,19 |
95 |
120 |
25 |
22,62 |
0,75 |
Test de corrélation de Spearman
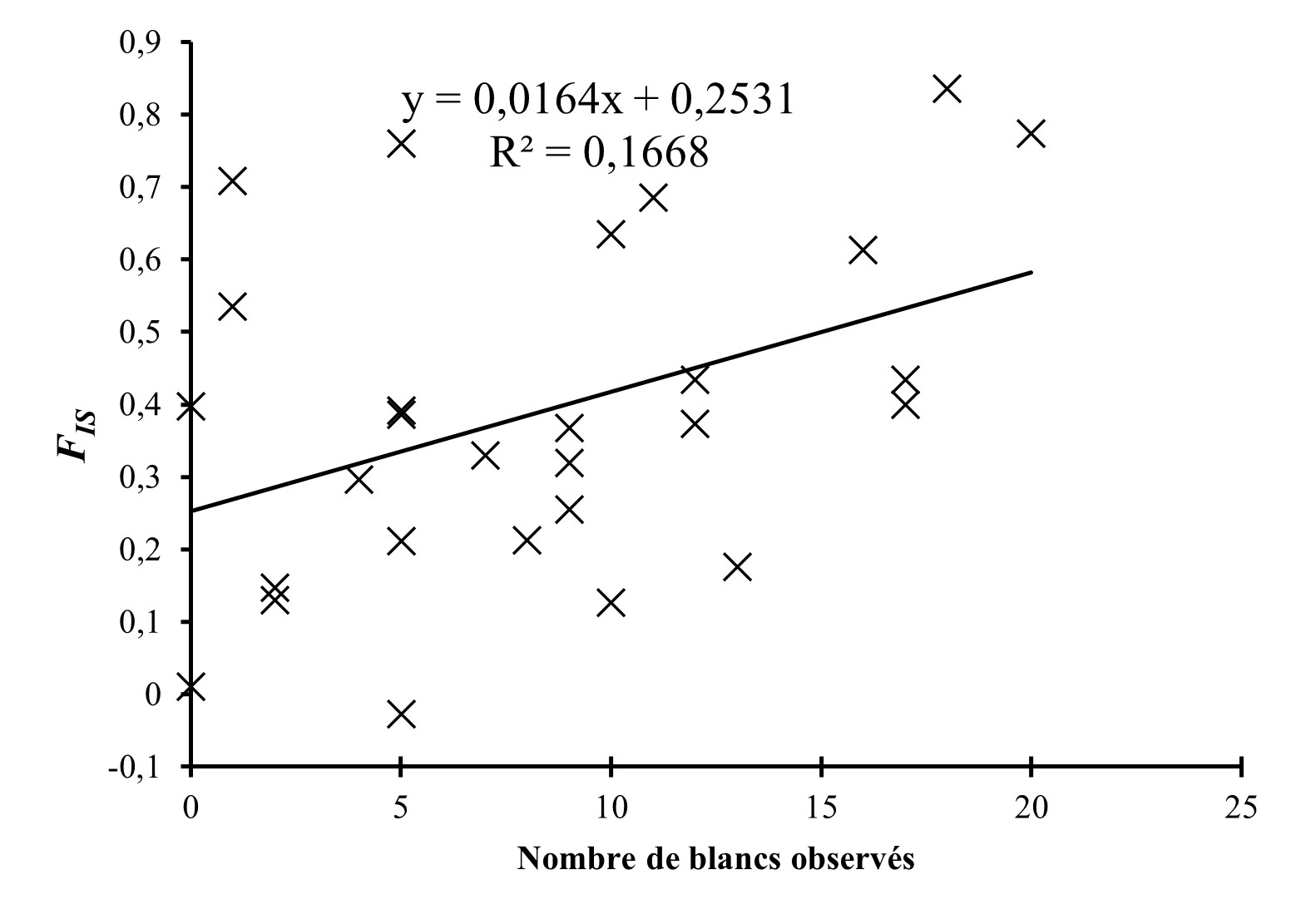
17Le test précédant a montré que les allèles nuls expliquent en partie les déficits en hétérozygotes. Pour estimer cette relation, la corrélation entre le nombre de blancs/locus/sous-population et l’Indice de Fixation (FIS) de Wright de chaque locus a été effectuée (figure 2). Celle-ci est ensuite analysée par un test de corrélation de Spearman. La figure 2 montre qu’il existe une relation positive forte entre ces deux variables avec une valeur de rho de Spearman = 0,372 (P-value = 0,0034). Le coefficient de détermination R² = 0,1668 montre qu’environ 16% des valeurs des FIS sont expliquées par les données manquantes.
18Figure 2 : Relation entre le nombre de génotypes blancs trouvés par locus et le FIS mesuré à ce locus sur l’ensemble des sites pour les 2 espèces de Clarias
Déséquilibre de liaison
19Le déséquilibre de liaison par paire de loci a été testé avec les 7 marqueurs microsatellites avec N (N-1)/2 combinaisons possibles, soit 21 paires pour sept loci étudiés pour l’ensemble des sous-populations des deux espèces par site. Sur les 21 combinaisons, aucun déséquilibre de liaison n’est significatif au niveau des deux espèces ce qui traduit une indépendance génétique entre les différents marqueurs étudiés (tableau 7).
Tableau 7 : Résultats des tests de déséquilibre de liaison par paire de loci dans chaque site et par espèce de Clarias
|
Paire de loci |
Clarias anguillaris |
Clarias gariepinus |
||||||
|
Bama |
Bala |
Kompienga |
Sourou |
Bama |
Bala |
Kompienga |
Sourou |
|
|
Cga01 x Cga03 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
Cga01 x Cga05 |
1 |
0,52 |
1 |
0,15 |
1 |
1 |
NA |
NA |
|
Cga01 x Cga06 |
1 |
1 |
1 |
0,08 |
1 |
1 |
0,97 |
1 |
|
Cga01 x Cga07 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
Cga01 x Cga09 |
1 |
1 |
0,49 |
1 |
0,15 |
1 |
0,08 |
1 |
|
Cga01 x Cga10 |
1 |
0,14 |
1 |
1 |
1 |
1 |
1 |
1 |
|
Cga03 x Cga05 |
0,12 |
0,3 |
1 |
1 |
0,08 |
1 |
1 |
NA |
|
Cga03 x Cga06 |
1 |
1 |
NA |
1 |
0,74 |
1 |
0,52 |
1 |
|
Cga03 x Cga07 |
0,19 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
Cga03 x Cga09 |
0,18 |
1 |
0,5 |
1 |
0,06 |
1 |
1 |
1 |
|
Cga03 x Cga10 |
0,5 |
1 |
1 |
0,19 |
0,26 |
1 |
1 |
1 |
|
Cga05 x Cga06 |
0,71 |
1 |
1 |
1 |
0,3 |
1 |
1 |
NA |
|
Cga05 x Cga07 |
0,29 |
0,25 |
1 |
1 |
0,63 |
1 |
1 |
NA |
|
Cga05 x Cga09 |
0,29 |
1 |
1 |
1 |
0,58 |
1 |
1 |
NA |
|
Cga05 x Cga10 |
0,12 |
1 |
1 |
1 |
0,26 |
1 |
1 |
NA |
|
Cga06 x Cga07 |
1 |
1 |
1 |
1 |
1 |
1 |
0,01 |
1 |
|
Cga06 x Cga09 |
1 |
1 |
NA |
1 |
0,24 |
1 |
0,48 |
1 |
|
Cga06 x Cga10 |
0,85 |
1 |
1 |
1 |
0,06 |
1 |
1 |
1 |
|
Cga07 x Cga09 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
Cga07 x Cga10 |
0,14 |
1 |
1 |
1 |
1 |
1 |
1 |
1 |
|
Cga09 x Cga10 |
1 |
1 |
1 |
1 |
0,39 |
1 |
1 |
1 |
Légende : NA = donnée manquante
Différenciation génétique entre les deux espèces
20Dans le tableau 8 sont inscrites les valeurs des FST entre les deux espèces avec les probabilités associées au niveau de chaque site. Le FST moyen est de 0,042 mais varie d’un site à un autre. La plus forte valeur a été observée sur le site du Sourou tandis que la plus faible a été constatée à Bala. Parmi les quatre tests, un seul est significatif et un autre marginalement significatif mais en combinant ces 4 tests selon la méthode binomiale généralisée, on obtient une P-value significative (P = 0,02). Ces résultats montrent que dans un même site, les poissons avec peu ou beaucoup de branchiospines sont génétiquement issus de populations différentes. Les différentes valeurs des FST traduisent une faible différentiation génétique entre les deux espèces.
Tableau 8 : Différentiation génétique entre les deux espèces de Clarias dans les 4 sites d’étude
|
Clarias anguillaris |
Clarias gariepinus |
FST |
P-value |
|
Bama |
Bama |
0,05 |
0,34 |
|
Bala |
Bala |
0,01 |
0,36 |
|
Kompienga |
Kompienga |
0,03 |
0,06 |
|
Sourou |
Sourou |
0,08 |
0,02 |
|
Sur l’ensemble |
0,04 |
0,02 |
|
Détection d’une structure cachée : effet Walhund
21Le tableau 9 présente les résultats des FIS dans le cas ou les deux espèces sont séparées et lorsqu’elles sont confondues sur l’ensemble des loci et dans chaque site. Ces résultats montrent un déficit important en hétérozygote chez la plupart des loci. Chez C. anguillaris, certains loci présentent des excès en hétérozygote, d’autres sont par contre fixés selon les sites. Cette remarque est également notée chez C. gariepinus. Les déficits observés sont plus important au niveau des loci Cga05 et Cga06. Ces déficits en hétérozygote pourraient se traduire par l’existence d’une structure cachée (effet Walhund). Pour tester l’existence d’une structure cachée le test de Wilcoson sur données appariées entre le FIS de chaque espèce prise séparement avec le FIS poolé des deux espèces a été réalisé. En supposant que les valeurs des FIS des espèces prises séparément sont inférieurs aux FIS des espèces poolées. Ainsi, les résultats des P-values des FIS de C. anguillaris et ceux poolés et entre C. gariepinus et ceux poolés donnent respectivement P = 0, 097 et P = 0, 074. Ces résultats montrent que les valeurs des FIS des espèces prises séparement sont marginalement inferieurs aux valeurs des FIS poolés et ne confirment donc pas que ces déficits proviennent d’un effet Walhund.
Tableau 9 : Résultats du FIS moyen par locus de Clarias anguillaris et Clarias gariepinus par espèce séparée et confondue
|
Sites |
Locus |
Clarias anguillaris |
Clarias gariepinus |
Poolé |
|
Bala |
Cga01 |
0,24 |
-0,14 |
0,21 |
|
Cga03 |
-0,06 |
0,18 |
-0,03 |
|
|
Cga05 |
0,41 |
0,2 |
0,39 |
|
|
Cga06 |
0,79 |
1 |
0,84 |
|
|
Cga07 |
0,31 |
0,22 |
0,3 |
|
|
Cga09 |
0,38 |
0,05 |
0,32 |
|
|
Cga10 |
0,2 |
-0,09 |
0,15 |
|
|
Bama |
Cga01 |
0,27 |
0,39 |
0,37 |
|
Cga03 |
0,28 |
0,58 |
0,54 |
|
|
Cga05 |
0,52 |
0,88 |
0,76 |
|
|
Cga06 |
0,65 |
0,7 |
0,71 |
|
|
Cga07 |
0,16 |
-0,04 |
0,01 |
|
|
Cga09 |
0,31 |
0,43 |
0,4 |
|
|
Cga10 |
0,2 |
0,12 |
0,13 |
|
|
Kompienga |
Cga01 |
1 |
0,09 |
0,43 |
|
Cga03 |
0 |
0,38 |
0,4 |
|
|
Cga05 |
0,7 |
0,81 |
0,77 |
|
|
Cga06 |
0 |
0,54 |
0,5 |
|
|
Cga07 |
0,22 |
0,28 |
0,26 |
|
|
Cga09 |
0 |
0,15 |
0,17 |
|
|
Cga10 |
0,25 |
0,42 |
0,37 |
|
|
Sourou |
Cga01 |
0,17 |
0,07 |
0,19 |
|
Cga03 |
0,2 |
-0,03 |
0,16 |
|
|
Cga05 |
0,6 |
NA |
0,68 |
|
|
Cga06 |
0,54 |
0,75 |
0,61 |
|
|
Cga07 |
0,44 |
0,22 |
0,39 |
|
|
Cga09 |
0,21 |
0 |
0,12 |
|
|
Cga10 |
0,45 |
0 |
0,33 |
Relation entre distance génétique et le nombre de branchiospines
Méthode de Mantel
22Les résultats du test de Mantel réalisé entre la distance génétique et celle due à la différence du nombre de branchiospines entre les individus d’un même site sont présentés dans le tableau 10. Pour l’ensemble des tests, un seul est significatif et en combinant les 4 tests selon la méthode binomiale généralisée l’on obtient une P-value non significative (P = 0,156). Ce résultat traduit l’absence de corrélation entre la distance génétique et le nombre de branchiospines. Les coefficients de détermination (R2) affichent des valeurs très faibles traduisant une quasi-absence de relation entre les deux variables.
Tableau 10 : Résultats des tests de corrélation entre la distance génétique et la distance de branchiospines chez les deux espèces de Clarias confondues
|
Sites |
P-value |
Coefficient de détermination R² |
|
Bala |
0,43 |
0 |
|
Bama |
0,59 |
5. 10-5 |
|
Sourou |
0,05 |
0,01 |
|
Kompienga |
0,18 |
0,01 |
|
Sur l'ensemble |
0,16 |
0,01 |
Méthode de régression
23La régression entre le nombre de branchiospines ainsi que les cordonnées génétiques des individus sur les 4 premiers axes de l’AFC, les facteurs environnementaux (longitude, latitude) ainsi que leurs interactions est présentée dans le tableau 11. Les résultats montrent que les cordonnées génétiques des individus sur l’axe 1 sont très fortement corrélées avec la distribution du nombre de branchiospines. L’axe 3 présente également une corrélation positive aux nombres de branchiospines. L’axe 4 est très marginalement significatif. Quant aux interactions, seulement celle de l’axe2-latitude est corrélée aux nombres de branchiospines. L’interaction entre l’axe4-longitude est très marginalement corrélée. Par contre, les facteurs environnementaux, pris individuellement, ne sont pas corrélés à la variabilité du nombre de branchiospines. Ces résultats montrent qu’il existe une forte corrélation entre le nombre de branchiospines et les coordonnées génétiques des individus dans l’espace (1, 2 et 3) de l’AFC.
Tableau 11 : Régression entre le nombre de branchiospines, les coordonnées génétiques des individus des 4 premiers axes chez les 2 espèces de Clarias confondues
|
Df |
Deviance Resid |
Df |
Resid. Dev |
F |
Pr (>F) |
||
|
Null |
96 |
23917 |
|||||
|
Axe1 |
1 |
3538,9 |
95 |
20378 |
18,32 |
0 |
*** |
|
Axe2 |
1 |
25,6 |
94 |
20353 |
0,13 |
0,72 |
|
|
Axe3 |
1 |
1077,7 |
93 |
19275 |
5,58 |
0,02 |
* |
|
Axe4 |
1 |
587,3 |
92 |
18688 |
3,04 |
0,08 |
. |
|
Latitude |
1 |
72,6 |
91 |
18615 |
0,38 |
0,54 |
|
|
Longitude |
1 |
0 |
90 |
18615 |
0 |
0,99 |
|
|
Axe2: latitude |
1 |
993,2 |
89 |
17622 |
5,14 |
0,03 |
* |
|
Axe4: longitude |
1 |
620,6 |
88 |
17001 |
3,21 |
0,08 |
. |
Les indices '***' ; '*' et '.' indiquent respectivement le seuil de signification à 0,001 ; 0,05 et 0,1.
Df : Degre of liberty ; Deviance Resid : Déviance résiduelle ; Resid. Dev : Variance résiduelle.
Relation entre le nombre de branchiospines et les facteurs environnementaux
24Les résultats de l’analyse de corrélation entre le nombre de branchiospines, les facteurs génétiques (espèce, les coordonnées génétiques des individus des 4 premiers axes), les facteurs environnementaux (site, longitude et la latitude) ainsi que leurs interractions sont résumés dans le tableau 12. L’analyse du tableau montre que parmis les facteurs génétiques, seulement le facteur espèce est très significativement (p<0,001) correlés au nombre de branchiospines. Les autres facteurs génétiques comme l’axe 3 et l’intéraction axe1 :axe4 sont corrélés au seuil de 5%. Parmi les facteurs environnementaux, seule l’espèce est très significativement (p<0,001) au nombre de branchiospines. Quant aux interractions entre les facteurs, on notre que l’intéraction espèce:site est très significativement corrélée à la variabilité du nombre de branchiospines. Le facteur espèce contribue à lui seul 87,22% de la variance totale, ce qui montre que le nombre de branchiospines est très pertinent pour discriminer les deux espèces
Tableau 12 : Analyse de régression entre le nombre de branchiospines, les facteurs génétiques et les facteurs environnementaux chez les 2 espèces de Clarias confondues.
|
Df |
Deviance Resid. |
Df |
Resid. Dev |
Pr (>Chi) |
||
|
NULL |
96 |
23917,3 |
||||
|
Espèce |
1 |
20860,1 |
95 |
3057,2 |
< 2,2e-16 |
*** |
|
Site |
3 |
399,6 |
92 |
2657,6 |
0 |
*** |
|
Axe1 |
1 |
0 |
91 |
2657,6 |
0,98 |
|
|
Axe2 |
1 |
4,8 |
90 |
2652,8 |
0,64 |
|
|
Axe3 |
1 |
88 |
89 |
2564,8 |
0,05 |
* |
|
Axe4 |
1 |
24,5 |
88 |
2540,3 |
0,29 |
|
|
Espèce:site |
3 |
536 |
85 |
2004,2 |
0 |
*** |
|
Axe1:Axe2 |
1 |
80,4 |
84 |
1923,9 |
0,06 |
|
|
Axe1:Axe4 |
1 |
100,1 |
83 |
1823,8 |
0,03 |
* |
Les indices '***' ; '*' et '.' indiquent respectivement le seuil de signification à 0,001 ; 0,05 et 0,1.
Df : Degré de liberté ; Deviance Resid : Déviance résiduelle ; Resid. Dev : Variance résiduelle.
Dendrogramme basé sur la méthode NJTRee
25La figure 3 présente les distances génétiques entre les différentes populations selon l’espèce et le site géographique. Elles ont été obtenues selon la méthode NJTRee (Neighbor-joining tree) sur les distances de corde de Cavalli-Sforza et Edwards. L’analyse de la figure fait ressortir deux groupes distincts. Le premier groupe est constitué des individus du site de Bala et le deuxième de ceux de Bama, Sourou et Kompienga. A l’intérieur de chaque site, les deux espèces sont distinctes. Mais l’échelle (0,05) temoigne une faible différentiation entre ces populations.
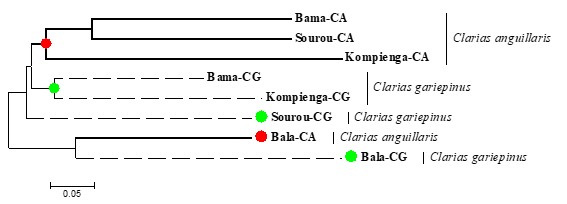
Figure 3 : Dendrogramme basé sur la méthode NJTRee entre Clarias gariepinus (vert) et Clarias anguillaris (rouge).
Discussion
26L’utilisation des 7 marqueurs retenus ont permis d’isoler et de caractériser l’ensemble des individus des deux espèces, bien que ces marqueurs aient été isolés initialement pour le génotypage de Clarias gariepinus (Galbusera et al., 1996). Ces marqueurs peuvent donc être utilisés sur les deux espèces. Ces marqueurs avaient déjà été utilisés pour caractériser d’autres espèces de la famille des Clariidae particulièrement chez Heterobranchus longifilis, Valenciennes, 1840 (Galbusera et al., 1996).
27Cependant, les analyses effectuées avec le logiciel Micro-checker et le test de corrélation de Spearman ont mis en évidence l’existence d’un nombre important d’allèles nuls sur l’ensemble des marqueurs. La présence d’allèles nuls a été signalée chez d’autres espèces de poissons notamment chez les Aloses (Rougemont, 2012). Les mutations ponctuelles sont en général à l’origine de ces allèles nuls. La non amplification d’un bon nombre d’individus lors de cette étude pourrait donc s’expliquer par ces mutations car leurs présences dans le génome rendent impossible l’appariement des amorces utilisées lors de la PCR comme cela a été montré déjà dans plusieurs travaux (Callen et al., 1993; Lehmann et al., 1998). Cette caractéristique rend parfois difficile l’interprétation des résultats obtenus avec les microsatellites.
28Le test de déséquilibre de liaison effectué montre qu’aucun déséquilibre de liaison n’a été observé chez aucune des deux espèces ce qui traduit une indépendance « statistique » des gènes. Les résultats obtenus peuvent donc être interprétés sans tenir compte d’une liaison possible des gènes.
29La différentiation génétique calculée à partir du FST entre paire de populations montre une différenciation faible entre les deux espèces. Ce résultat est connfirmé par les distances génétiques obtenues par la méthode des distances de corde de Cavalli-Sforza et Edwards. Cette proximité génétique des deux espèces avait déjà été signalée par Teugels et al. (1992) et pourrait traduire l’existence d’un flux génétique important. Etant donné que ces deux espèces vivent en sympatrie dans la plupart des plans d’eau. Ce flux génétique entre ces deux espèces pourrait provenir probablement du phénomène d’hybridation qui a lieu en milieu naturel. Ce phénomène est très connu chez les espèces de la famille des Clariidae donnant parfois des descendants viables et fertiles. C’est le cas par exemple de l’hybridation entre C. gariepinus et C. macrocephalus (Gunther, 1864) très pratiquée dans les systèmes d’élevages en Asie (Senanan et al., 2004). Un autre phénomène que pourrait expliquer cette faible différenciation génétique est un taux de mutation très faible entre deux espèces (Simard, 1999), mais ce qui ne semble pas être le cas dans cette étude au regard du fort taux des allèles nuls imputable aux mutations. Lorqu’on applique le temps de divergence selon la méthode de Ravel et al. (2013), qui suppose une absence de flux de gènes entre les deux espèces, on obtient 10 générations, ce qui est très faible. Par contre, en supposant qu'un flux de gène existe toujours et que les populations sont à l'équilibre de Hardy-Weinberg, il est possible d’utiliser la valeur attendue pour les deux populations comme proposé par Solano et al. (2009), ce qui donne un taux de migration m~0,022 entre les deux espèces par génération. Cela se fonde sur une hypothèse que les deux taxons étaient bien séparés et sont de nouveau en contact depuis peu.
30Le calcul des FIS a révélé l’existence de déficits important en hétérozygotes chez la plupart des loci et plus prononcé au niveau de Cga05 et Cga06. Un déficit important en hétérozygotes aux loci Cga05 et Cga09 avait été signalé par Galbusera et al. (1996). Ces déficits sont en général dus à l’effet Walhund, à la consanguinité, à la sélection (élimination d’un génotype particulier) et la non-détection du polymorphisme (allèles nuls, artefacts de lecture des gels). Les résultats sur le test de Wilcoxon ne révèlent pas l’existence d’une structure cachée (effet Walhund) c'est-à-dire la présence de deux espèces. L’origine donc de ces déficits pourrait provenir des autres facteurs tels la consanguinité, la sélection et les problèmes techniques. Pour ce qui est du cas des loci Cga05 et Cga06 où les déficits en hétérozygotes sont très prononcés, des contraintes spécifiques pourraient être à l’origine.
31Le nombre de branchiospines comme marqueur de différenciation entre les deux espèces a été testé par deux méthodes que sont : le test de Mantel et la méthode de régression. Les résultats du test de Mantel ne montrent pas une corrélation entre la distance génétique et celle du nombre de branchiospines. En revanche, les résultats obtenus avec la méthode de régression montrent que les coordonnées génétiques des individus de l’AFC sont fortement corrélées aux nombres de branchiospines de chaque espèce. Ce qui dénote que les données microsatellites permettent de discriminer les deux espèces sur la base du nombre de branchiospines.
32L’analyse de régression faite, le nombre de branchiospines, les facteurs génétiques et les facteurs environnementaux révèlent que la distribution des branchiospines est fortement corrélée aux facteurs génétiques (87%), ce qui montre que le génotype a un impact significatif sur le niveau d’expression des branchiospines. Et parmi les facteurs environnementaux (longitude, latitude, site), seul le facteur site explique en partie la variabilité du nombre de branchiospines. Ces données montrent que la distribution des branchiospines provient donc de l’interaction génotype-environnement. Cette interaction génotype-environnement d’un certain nombre de caractères a été déjà observée chez la carpe (Moa et al., 1975) et le saumon (Guillaume et al., 2007).
33En définitive, parmi les méthodes employées pour différencier les deux espèces de Clarias, seule la méthode de régression s’est montrée concluante. Quant aux tests de Mantel et l’effet Walhund, ils n’ont pas présentés de résultats probants.
Conclusion
34La caractérisation génétique de Clarias anguillaris et C. anguillaris à l’aide des marqueurs microsatellites a révélé une faible différenciation génétique entre ces deux espèces. Les résultats obtenus avec la méthode de régression montrent que les données microsatellites ont permis de discriminer les deux espèces. Mais reste cependant limité avec les autres méthodes que sont : le test de Mantel et l’effet Walhund. Quant à la question de savoir si la variabilité du nombre de branchiospines suit celle de l’environnement, l’étude montre que la distribution des branchiospines est expliquée en grande partie par les facteurs génétiques. Cependant, d’autres études utilisant d’autres marqueurs comme les SNP pourraient être utilisées pour avoir plus de précision sur la différenciation génétique des deux espèces.
Bibliography
Agnèse JF, Teugels GG, Galbusera P, Guyomard R et Volckaert F, 1997: Morphometric and genetic characterisation of sympatric populations of clarias gariepinus and c. Anguillaris from senegal. Journal of Fish Biology, 50: 1143-1157.
Belkhir K, Borsa P, Chikhi L, Raufaste N et Bonhomme F, 2004: Genetix 4.05, logiciel sous windows tm pour la génétique des populations, edited by P Laboratoire Génome, Interactions, CNRS UMR 5000, université de Montpellier II, Montpellier (France).
Benech V, Teugels G.G, Gourène G, 1993 : Critère pratique pour distinguer deux poissons-chats africains, Clarias anguillaris et C. gariepinus (Siluriformes; Clariidae). Cybium,17(1) pp 83-85
Boisneau P, 1990: Migration, répartition, reproduction et taxonomie des aloses (alosa, sp.) dans le bassin de la loire. Thèse de Docteur en sciences. Université Paris XII, 106 p.
Bonhomme F, Naciri M, Bahri-Sfar L et Lemaire C, 2002: Analyse comparée de la structure génétique de deux espèces de poissons marins apparentées et sympatriques dicentrarchus labrax et dicentrarchus punctatus. Comptes Rendus Biologies, 325: 213–220.
Callen DF, Thompson AD, Shen Y, Phillips H, Richards RI et al., 1993: Incidence and origin of "null" alleles in the (ac)n microsatellite markers. American Journal of Human Genetics, 52: 922-927.
Cavalli-Sforza LL et Edwards AWF, 1967: Phylogenetic analysis: Model and estimation procedures. American Journal of Human Genetics, 19: 233-257.
Chapuis MP et Estoup A, 2007: Microsatellite null alleles and estimation of population differentiation. Molecular Biology and Evolution, 24: 621-631.
Chesser RK et Nei M, 1983: Estimation of fixation indices and gene diversities. Annals of Human Genetics, 47: 253-259.
Compaoré I, Toguyéni A, Rougeot C, Kestemont P et Mélard C, 2015: Morphometric and meristic identification of wild populations of clarias sp and their hydro-geographical structuring in burkina faso. American Journal of Agricultural Science, 2: 208-216.
De Graaf G et Janssen J, 1996: Handbook on the artificial reproduction and pond rearing of the african catfish clarias gariepinus in sub-saharan africa, edited by FAO, Rome, 109p.
De Meeûs T, 2012: Initiation à la génétique des populations naturelles: Applications aux parasites et à leurs vecteurs, Marseille, ???p.
De Meeûs T, Guégan J F et Teriokhin A, 2009: Multitest v.1.2, a program to binomially combine independent tests and performance comparison with other related methods on proportional data. BMC Bioinformatics, 10 (1): 443 p..
Devaere S, Jansen G, Adriaens D et Weekers P, 2007: Phylogeny of the african representatives of the catfish family clariidae (teleostei, siluriformes) based on a combined analysis: Independent evolution towards anguilliformity. Journal of Zoological Systematics and Evolutionary Research, 45 (3): 214-229.
Ebrahimi M, Farajpour M et Rahimmalek M, 2012: Inter- and intra-specific genetic diversity of iranian yarrow species achillea santolina and achillea tenuifolia based on issr and rapd markers. Genetics and Molecular Research, 11 (3): 2855-2861.
Ellegren H, 2004: Microsatellites: Simple sequences with complex evolution. Nature Reviews Genetics, 5: 435-445.
Fritzner NG, Hansen MM, Madsen S et Kristiansen K, 2001: Use of microsatellite markers for identification of indigenous brown trout (salmo trutta l.) in a geographical region heavily influenced by stocked domesticated trout. Journal of Fish Biology, 58: 1197-1210.
Galbusera P, Volckaert AF, Hellemans B et Ollevier F, 1996: Isolation and characterization of microsatellite markers in the african catfish clarias gariepinus (burchell, 1822). Molecular Ecology, 5: 703-705.
Gibson RM, Pires D, Delaney KS et Wayne RK, 2005: Microsatellite DNA analysis shows that greater sage grouse leks are not kin groups. Molecular Ecology, 14: 4453-4459.
Goudet J, 2003: Fstat (version 2.9.4), a program (for windows 95 and above) to estimate and test population genetics parameters. Available from http://www.Unil.Ch/izea/softwares/fstat.Html updated from goudet (1995).
Goudet J, Raymond M, De Meeûs T et Rousset F, 1996: Testing differentiation in diploid populations. Genetics, 144: 1933-1940.
Guillaume C, Guy P, Pierre B et Louis B, 2007: The influence of gene-environment interactions on ghr and igf-1 expression and their association with growth in brook charr, salvelinus fontinalis (mitchill). BMC Genetics, 8 : 87 p.
Guo BY, Xie CX, Qi LL, Qi PZ, Wu CW et al., 2010: Assessment of the genetic diversity among glyptosternum maculatum, an endemic fish of yarlung zangbo river, tibet, china using ssr markers. Biochemical Systematics and Ecology, 38: 1116-1121.
Hecht T, 2013: A review of on-farm feed management practices for north african catfish (clarias gariepinus) in sub-saharan africa. In m.R. Hasan and m.B. New, eds. On-farm feeding and feed management in aquaculture. FAO Fisheries and Aquaculture Technical Paper No. 583. Rome, FAO.: 463–479.
Lehmann T, Hawley WA, Grebert H et Collins FH, 1998: The effective population size of anopheles gambiae in kenya: Implications for population structure. Molecular Biology and Evolution, 15 (3): 264-276.
Lévêque C, Paugy D, Teugels GG, 1992 : Faune des poissons d'eaux douces et saumâtres de l'Afrique de l’Ouest : tome 1, The Fresh and Brackish Water Fishes of West Africa VOLUME 2. Musée Royal de l'Afrique Centrale Tervuren, Belgique. 521p
Marchal E, 1964: Note sur deux caractères de sardinella aurita (c et v) de côte d'ivoire. Communication présenté au Conseil International pour l'Exploitation de la Mer (C.I.P.E.M).
Moa VR, Hulat AG et Wohlfart HG, 1975: Genetic differences between the chinese and european races of the common carp. I. Analysis of genotype-environment interactions for growth rate. Heredity, 34: 323-340.
Mohindra V, Singh RK, Palanichamy M, Ponniah AG et Lal KK, 2007: Genetic identification of three species of the genus clarias using allozyme and mitochondrial DNA markers. Journal of Applied Ichthyology, 23: 104-109.
Palsboll PJ, Bérubé M et Allendorf FW, 2006: Identification of management units using population genetic data. Trends in Ecology and Evolution, 22: 11-16.
Paugy D, Lévêque C et Teugels GG, 2003: Faune des poissons d'eaux douces et saumâtres de l'afrique de l'ouest : Tome 1 = the fresh and brackish water fishes of west africa : Volume 1. IRD, Paris (FRA) ; Tervuren, ???p.
Ravel J, Brotman RM, Gajer P, Ma B, Nandy M et al., 2013: Daily temporal dynamics of vaginal microbiota before, during and after episodes of bacterial vaginosis. Microbiome, : 1-29
Renshaw MA, Saillant E, Bradfield SC et Gold JR, 2006: Microsatellite multiplex panels for genetic studies of three species of marine fishes: Red drum (sciaenops ocellatus), red snapper (lutjanus campechanus), and cobia (rachycentron canadum). Aquaculture, 253: 731-735.
Rognon X, Teugels GG, Guyomard R, Galbusera P, Andriamanga M et al., 1998: Morphometric and allozyme variation in the african catfishes clarias gariepinus and c. Anguillaris. Journal of Fish Biology, 53: 192-207.
Rougemont Q, 2012: Etude de la différenciation et de la dispersion des populations des deux espèces d’aloses (alosa spp) en france : Apport de la génétique des populations et de la microchimie des otolithes. Mémoire d'Ingenieur. AGROCAMPUS OUEST, Rennes, 49 p.
Rukera Tabaro S, Micha JC et Ducarme C, 2005: Essais d’adaptation de production massive de juvéniles de clarias gariepinus en conditions rurales. Tropicultura, 23 (4): 231-244.
Schwartz MK, Luikart G et Waples RS, 2006: Genetic monitoring as a promising tool for conservation and management. Trends in Ecology and Evolution, 22: 25-33.
Senanan W, Kapuscinski AR, Na-Nakorn U et Miller LM, 2004: Genetics impacts of hybrid catfish farming (clarias macrocephalus x c. Gariepinus) on native catfish populations in central thailand. Aquaculture, 235: 167-184.
Simard F, 1999: Variablité génétique et flux de gènes chez les moustiques anopheles arabiensis et anopheles gambiae, vecteurs du paludisme en afrique. Apport des loci microsatellites. Université Henri Poincaré, Nancy-I, Nancy, 270 p.
Solano P, Ravel S, Bouyer J, Camara M, Kagbadouno MS et al., 2009: The population structure of glossina palpalis gambiensis from island and continental locations in coastal guinea. PLoS Neglected Tropical Diseases, 3 e392, ??p.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M et al., 2011: Mega5 : Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 28: 2731-2739.
Teriokhin AT, De Meeûs T et Guegan JF, 2007: On the power of some binomial modifications of the bonferroni multiple test. Zhurnal Obshchei Biologii, 68: 332-340.
Teugels GG, 1982: Preliminary results of a morphological study of five nominal species of the subgenus clarias (pisces: Clariidae). Journal of Natural History, London 16: 439-464.
Teugels GG, 1986: A systematic revision of the african species of the genus clarias (pisces; clariidae). Annales du Musée Royal de l’Afrique Centrale, 247: 1-199.
Teugels GG, Guyomard R et Legendre M, 1992: Enzymatic variation in african clariid catfishes. Journal of Fish Biology, 40: 87-96.
Van Oosterhout C, Hutchinson WF, Wills DPM et Shipley P, 2004: Micro-checker: Software for identifying and correcting genotyping errors in microsatellite data. Molecular Ecology Notes, 4: 535-538.
Weir BS et Cockerham CC, 1984: Estimating f-statistics for the analysis of population structure. Evolution, 38: 1358-1370.
Wong LL, Peatman E, Lu J, Kucuktas H, He S et al., 2011: DNA barcoding of catfish: Species authentication and phylogenetic assessment. PLoS ONE, 6: e17812.
Yu SCS et Quilang JP, 2014: Molecular phylogeny of catfishes (teleostei: Siluriformes) in the philippines using the mitochondrial genes coi, cyt b, 16s rrna, and the nuclear genes rag1 and rag2. Philippine Journal of Science, 143: 187-198.
To cite this article
About: Inoussa Compaoré*
Laboratoire de Recherche et de Formation en Pêche et Faune (LaRFPF)/Université Nazi BONI, 01 BP 1091 Bobo-Dioulasso 01, Burkina Faso
About: Aboubacar Toguyéni
Unité de Recherche en Aquaculture et Biodiversité Aquatique/Laboratoire d’Etude et de Recherche sur les Ressources Naturelles et Sciences de l’Environnement (UR-ABAQ/LERNSE), Université Nazi BONI, 01 BP 1091 Bobo-Dioulasso 01, Burkina Faso
About: Vincent Gennotte
Centre de Formation et de Recherche en Aquaculture (CEFRA), Université de Liège; 10, chemin de la justice, B-4500 Tihange, Belgique
About: Johan Michaux
Université de Liège : Département des sciences de la vie : Génétique et physiologie des microalgues
About: Carole Rougeot
Centre de Formation et de Recherche en Aquaculture (CEFRA), Université de Liège; 10, chemin de la justice, B-4500 Tihange, Belgique
About: Patrick Kestemont
Research Unit in Environmental and Evolutionary Biology, Université de Namur; Rue de Bruxelles 61-5000; Namur Belgique
About: Charles Mélard
Centre de Formation et de Recherche en Aquaculture (CEFRA), Université de Liège; 10, chemin de la justice, B-4500 Tihange, Belgique