Etude comparative de la diversité et de la structure génétique de populations sauvages de Phaseolus lunatus L. à l’aide des marqueurs enzymatiques et microsatellites
Reçu le 23 juin 2004, accepté le 11 février 2005
Résumé
La diversité génétique intra- et interpopulation de Phaseolus lunatus L. var. silvester originaire de la vallée centrale du Costa Rica a été étudiée à l’aide des marqueurs enzymatiques et microsatellites. Comme attendu, les marqueurs microsatellites ont montré une diversité génétique plus élevée que les marqueurs enzymatiques. Toutefois, la diversité génétique mise en évidence demeure modérée chez le haricot de Lima et pourrait être liée à l’origine des échantillons restreinte à la vallée centrale du Costa Rica ou à l’origine des amorces microsatellites. La diversité génétique totale (HT), la diversité génétique intrapopulation (HS) sont sous-estimées avec les marqueurs enzymatiques. La contribution de la diversité génétique interpopulation (DST) à la diversité génétique totale est identique avec les deux types de marqueurs. Les marqueurs enzymatiques et microsatellites révèlent un haut niveau de consanguinité pour l’ensemble des populations (FIT) et pour les sous-populations (FIS). La diversité génétique intrapopulation (HS) représente 70 % avec les deux types de marqueurs génétiques. Ainsi, les populations individuelles du haricot de Lima dans la vallée centrale du Costa Rica peuvent constituer des unités génétiques valables dans le cadre d’une conservation in situ. Les sites naturels des populations sauvages sont sous l’effet d’un changement accéléré de la vocation des terres à cause de la pression démographique et de l’intensification de l’agriculture. Il importe de mener une conservation ex situ couplée avec une conservation in situ. Les populations les plus menacées de disparition devront faire l’objet de collectes de graines pour une conservation ex situ.
Abstract
Comparative study of genetic diversity and structure of wild Phaseolus lunatus L. populations using enzymatic and microsatellite markers. Genetic diversity within and among Phaseolus lunatus L. var. silvester populations from the central valley of Costa Rica was studied using enzymes and microsatellite markers. As expected, microsatellite markers showed more genetic diversity than enzymes markers. However, the relatively moderate genetic diversity displayed can be explained by restricted origin of samples to the central valley of Costa Rica or by the source of the primer. Total genetic diversity (HT) and within population genetic diversity (HS) were underestimated with enzyme markers. The contribution of among population genetic diversity (DST) to total genetic diversity was similar for both markers. Enzymes and microsatellite markers pointed out a high inbreeding level for whole population (FIT) and within population (FIS). Within population genetic diversity represents 70% for both genetic markers. So, Lima bean wild populations at the central valley of Costa Rica may constitute valid in situ conservation unit. Wild populations natural sites are submitted to an accelerate change of land use because of demographic pressure and agriculture intensification. It is necessary to conduct an ex situ conservation associated with an in situ conservation. Seeds from the most threatened populations should be collected for an ex situ conservation.
1. Introduction
1Le haricot de Lima, Phaseolus lunatus L., est originaire d’Amérique latine, où la forme sauvage se présente comme une herbacée annuelle ou pluriannuelle, caractérisée par un habitus de croissance indéterminé volubile et par une allogamie facultative. L’espèce est cultivée de nos jours sur les continents africain, américain et asiatique. Une classification infra spécifique distingue les formes sauvages P. lunatus var. silvester, des formes cultivées, P. lunatus var. lunatus (Baudet, 1977). Au Costa Rica, de nombreuses populations sauvages du haricot de Lima sont localisées dans un haut plateau dénommé vallée centrale et à des altitudes comprises entre 800 et 1500 m. La vallée centrale du Costa Rica est une zone située entre les chaînes montagneuses de la cordillère centrale au nord et de Talamanca au sud. Cette vallée s’étend sur 2000 km2 environ, formant une bande de 80 km de long sur 30 km de large. Le premier recensement de populations effectué en 1997 y a dénombré 410 populations (Lodieu, 1997). La vallée centrale du Costa Rica est considérée comme une région de diversité pour la forme sauvage du haricot de Lima dont les populations sont menacées par la destruction de leurs habitats naturels (Rocha et al., 1997). Ces auteurs ont évalué le taux d’extinction annuel des populations à 30 %. La destruction des sites naturels par l’aménagement des terrains, des caféières et la destruction des haies localisées dans les zones urbaines et agricoles, de même que le contrôle des adventices suite à l’intensification de l’agriculture contribuent ensemble à 89,5 % des cas de disparition de populations du haricot de Lima (Rocha et al., 1997). Cela a conduit à la mise en place d’un projet de conservation in situ avec la collaboration de l’International Plant Genetic Resources Institute (IPGRI), de la Faculté universitaire des Sciences agronomiques de Gembloux (Belgique) et de l’Université du Costa Rica.
2Plusieurs études antérieures menées dans le cadre de ce projet sur les populations sauvages du haricot de Lima ont permis de connaître
3– le déterminisme génétique des marqueurs enzymatiques dans le pool génique primaire de P. lunatus (Zoro Bi et al., 1999),
4– la taille optimale des échantillons de graines à prélever par population et le nombre de populations nécessaire pour retenir toutes les combinaisons d’allèles enzymatiques (Zoro Bi et al., 1998),
5– les conséquences du système de reproduction de la forme sauvage du haricot de Lima sur la conservation des graines (Degreef et al., 1997),
6– le flux de gènes direct en rapport avec les distances de vol de Apis mellifera L., principal pollinisateur des populations, l’aire moyenne de voisinage et la taille efficace des populations de P. lunatus (Hardy et al., 1997) et
7– la démographie des populations synthétiques de P. lunatus conservées in situ (Meurrens et al., 2001).
8Les premières analyses de la variabilité génétique des populations sauvages du haricot de Lima dans la vallée centrale ont été réalisées avec l’aide de marqueurs enzymatiques. Les marqueurs enzymatiques présentent plusieurs avantages et inconvénients (Murphy et al., 1990 ; Buth, Murphy, 1999 ; Bank et al., 2001). Au titre des avantages, ils ont un pouvoir de discrimination plus élevé que les marqueurs morphologiques, ils sont utilisés comme marqueurs pour les gènes qui les codent, sont neutres et peuvent être révélés dans plusieurs organes à différents stades de développement. Toutefois, la faiblesse du nombre moyen d’allèles, l’occurrence de mutations silencieuses et d’allèles nuls, l’expression de certains enzymes en fonction du stade de développement et la variabilité génétique limitée aux loci codants constituent des désavantages.
9Ces limites des marqueurs enzymatiques ont justifié la recherche de marqueurs plus polymorphes comme les microsatellites. Ces derniers sont co-dominants, comme les marqueurs enzymatiques, mais ils possèdent un pouvoir de discrimination souvent plus élevé (Stachel et al., 2000 ; Fahima et al., 2002 ; Ford et al., 2002). Afin de ne pas procéder à l’étape coûteuse d’isolement des amorces microsatellites propres à l’espèce P. lunatus, des tests d’amplification croisée à partir d’amorces isolées chez P. vulgaris L. par le Centro Internacional de Agricultura Tropical (CIAT) ont été conduits.
10La présente étude a pour objectif d’évaluer comparativement la variabilité et la structure génétiques des populations sauvages du haricot de Lima à l’aide des marqueurs enzymatiques et microsatellites. Les résultats de cette évaluation peuvent contribuer à la conservation in situ de la légumineuse dans une région de diversité.
2. Matériel et méthodes
2.1. Matériel
11Vingt-huit populations sauvages de P. lunatus provenant de différentes régions de la vallée centrale du Costa Rica et collectées en 1998 ont été caractérisées à l’aide des marqueurs enzymatiques. Seules des populations contenant au minimum quatre individus ont été retenues. Dix graines prélevées de manière aléatoire parmi les individus de chaque population ont été analysées.
12Pour ce qui est du matériel étudié à l’aide des marqueurs microsatellites, nous avons choisi neuf populations originaires de la région d’Heredia, choisie pour le nombre élevé de populations sauvages de la légumineuse : 359 individus, chacun représenté par un échantillon de feuilles séchées au silicagel, ont été collectés en 2000 et 2001. Pour l’échantillonnage, nous sommes partis d’une population centrale (composée de plus de 50 individus), et toutes les populations incluses dans un rayon de 3 km autour de cette population ont été retenues pour le prélèvement des feuilles (Ouédraogo, 2003).
2.2. Méthodes
13Les procédures d’extraction des enzymes, d’électrophorèse sur gel d’amidon et de révélation des loci enzymatiques ont été mises au point lors de travaux antérieurs (Maquet, 1995 ; Maquet et al., 1997 ; Zoro Bi et al., 1997 ; Zoro Bi et al., 1999). Treize systèmes enzymatiques (ADH, DIA, END, CEST, G6PDH, GDH, GPI, LAP, MDH, PGDH, PGM, SKDH et SOD) ont permis de caractériser la diversité génétique des échantillons de graines collectés dans la vallée centrale, révélant 25 loci enzymatiques.
14L’utilisation des marqueurs microsatellites a nécessité la mise au point d’un protocole d’extraction de l’ADN à partir de feuilles séchées au silicagel, des tests d’amplification croisée de 73 couples d’amorces microsatellites isolées par le CIAT chez P. vulgaris, des tests de polymorphisme de 57 couples d’amorces et le choix de 10 couples d’amorces microsatellites générant des profils polymorphes parmi le matériel collecté.
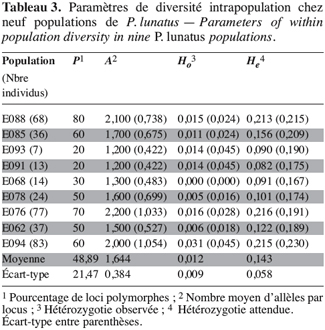
15Extraction de l’ADN. L’extraction de l’ADN total des échantillons de P. lunatus a été réalisée selon un protocole mis au point par Ouédraogo (2003) : il est simple, rapide, et nécessite de petites quantités de matériel végétal (80 à 100 mg de feuilles séchées au silicagel). Le tampon d’extraction est composé de : 0,25 M pH 8 Tris ; 2,1 M NaCl ; 3 % CTAB : 0,05 M EDTA et de 0,2 % de ß-mercaptoéthanol. Le protocole permet d’atteindre un rendement régulier d’au minimum 15 mg d’ADN/g de feuilles séchées. Les fragments d’ADN sont de bonne qualité et ont été restreints avec succès avec l’endonucléase ECOR I. Ce protocole est également efficace sur des feuilles séchées au silicagel de P. vulgaris, P. coccineus L. et Cajanus cajan L.
16Amplification des loci microsatellites de P. lunatus et électrophorèse. L’amplification de l’ADN a été faite avec 10 marqueurs microsatellites (AG1, BM98, BM141, BM142, BM149, BM156, BM160, BM161, GATS54 et GATS91) isolés chez P. vulgaris. Les amorces microsatellites ont été synthétisées par la firme Eurogentec et la Taq DNA polymerase a été fournie par Amersham Pharmacia Biotech de même que les dNTPs. Les amplifications PCR ont été réalisées à l’aide d’un Peltier Thermal Cycler 200 de MJ Research. L’amplification a été faite avec 20 ng d’ADN total et dans un volume final de 20 mL contenant un tampon d’amplification d’une concentration de 1x, des amorces à une concentration de 0,1 mM, des dNTPs à 0,25 mM et 1,5 U de Taq DNA polymérase. Les programmes d’amplification varient en fonction des amorces.
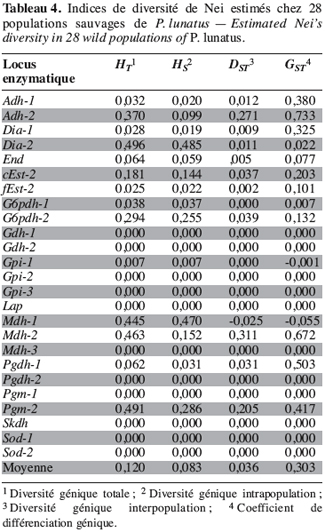
17L’électrophorèse a été faite à l’aide de cuves de séquençage (Model S2 de GIBCO BRL Life Technologies) et le gel de polyacrylamide a été soumis à une électrophorèse préliminaire d’une heure (1800 V, 40 mA et 80 W) dans un tampon continu de TBE 1x. L’électrophorèse proprement dite a été réalisée dans les mêmes conditions (voltage, intensité et puissance) que l’électrophorèse préliminaire durant 1 h à 1 h 30 et ce, après avoir chargé 5 mL dans les puits du gel.
18Pour la révélation des profils, le gel de polyacrylamide après l’électrophorèse est fixé dans une solution composée d’alcool éthylique absolu (10 %) (Vel 1115) et d’acide acétique (0,5 %) (Vel 1005) pendant 7 mn. Il est ensuite traité dans une solution aqueuse contenant 0,1 % de nitrate d’argent (Aldrich cat. 20,913-9) pendant 7 mn. Le gel est enfin lavé (1 s) dans de l’eau désionisée et les profils générés par les couples d’amorces microsatellites sont révélés dans une solution de développement (1,5 % NaOH, 0,2 % formaldéhyde).
19Les gels de polyacrylamide ont été archivés sur support informatique (disquette) grâce à un système d’imagerie digitale. Les images en format TIFF ont été transférées sur PC pour la conversion des fichiers images dans le logiciel Gelcompar ver. 4.2 (Applied Maths., 1997) et la détermination de la taille des fragments amplifiés des différents échantillons.
20Analyse des données. Le pourcentage de loci polymorphes (P), le nombre moyen d’allèles par locus (A), l’hétérozygotie observée (Ho) et l’hétérozygotie attendue (He) ont été obtenus avec POPGENE ver. 1.32 (Yeh, Boyle, 1997). La diversité génétique totale (HT), la diversité génétique intrapopulation (Hs), la diversité génétique interpopulation (DST) et le coefficient de différenciation entre les populations, dénommé aussi coefficient de différenciation génique (GST) ont été obtenus avec le logiciel FSTAT ver. 2.9.3.2. (Goudet, 1995). Pour rappel, la diversité génétique totale est la somme de la diversité génétique intrapopulation et de la diversité génétique interpopulation (Crossa et al., 1993). Le coefficient de différenciation génique (GST) traduit la proportion de la diversité génétique totale due à la variabilité interpopulation. Les F-statistiques de Wright permettent aussi de connaître la structure génétique d’une espèce grâce à l’estimation, d’une part, de la fixation des allèles dans l’ensemble des populations (FIT) et à l’intérieur des sous-populations (FIS) et, d’autre part, de la différenciation génétique entre les sous-populations (FST).
3. Résultats
3.1. Diversité génétique
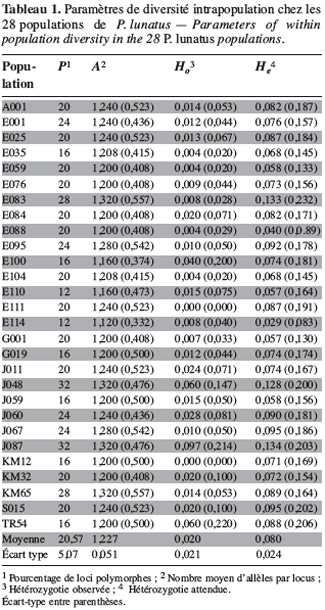
21Marqueurs enzymatiques. La plupart des loci enzymatiques sont dialléliques. Pour l’ensemble des populations étudiées, le seuil de polymorphisme a été fixé à 5 %. Les fréquences alléliques mettent en évidence 18 loci monomorphes (Adh-1, Dia-1, End, fEst-2, Gdh-1, Gdh-2, Gpi-1, Gpi-2, Gpi-3, G6pdh-1, Lap, Mdh-3, Pgdh-1, Pgdh-2, Pgm-1, Skdh, Sod-1 et Sod-2) et sept loci polymorphes (Adh-2, Dia-2, cEst-2, G6pdh-2, Mdh-1, Mdh-2 et Pgm-2). A partir des fréquences alléliques, les indices de polymorphisme intrapopulation ont été déterminés pour l’ensemble des 28 populations sauvages de P. lunatus. Le tableau 1 regroupe les paramètres de diversité génétique intrapopulation de l’échantillon étudié.
22Les indices de polymorphisme intrapopulation P, A, Ho et He sont caractérisés par une forte hétérogénéité. Le nombre moyen d’allèles par locus (A) est peu élevé chez les populations analysées. L’hétérozygotie moyenne observée par population (Ho = 0,020 ± 0,021) est faible et inférieure à l’hétérozygotie moyenne attendue par population dans les conditions de Hardy-Weinberg (He = 0,080 ± 0,024) (Tableau 1).
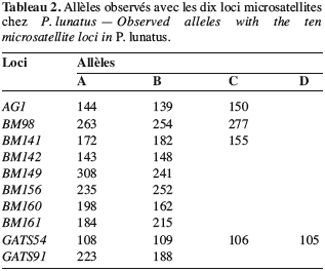
23Marqueurs microsatellites. Les génotypes des populations sauvages sont caractérisés à partir de 10 loci microsatellites codant pour 25 allèles. Le tableau 2 montre les loci microsatellites et les allèles correspondants dont la taille est exprimée en paires de base.
24Les mêmes paramètres de diversité génétique intrapopulation ont été calculés (Tableau 3). Les indices de polymorphisme intrapopulation sont également hétérogènes, mais les valeurs obtenues avec les marqueurs microsatellites sont en général supérieures à celles observées avec les marqueurs enzymatiques. Toutefois, on constate toujours un nombre peu élevé d’allèles par locus (A).
25L’hétérozygotie moyenne observée par population (Ho = 0,012 ± 0,009) est plus faible que celle obtenue avec les marqueurs enzymatiques. Elle est aussi inférieure à l’hétérozygotie moyenne attendue (He = 0,143 ± 0,058), laquelle est par contre 1,8 fois plus élevée que celle obtenue avec les marqueurs enzymatiques.
3.2. Structure génétique
26Marqueurs enzymatiques. Les indices de diversité génétique de Nei ont été estimés pour l’ensemble des 28 populations (Tableau 4). La diversité génétique totale (HT) et la diversité génétique intrapopulation (HS) sont respectivement de 0,120 et de 0,083. Il existe une grande divergence génétique entre les populations de P. lunatus. En effet, la contribution de la diversité génétique interpopulation (GST) à la diversité génétique totale est en moyenne de 30 %.
27Les valeurs élevées du coefficient de consanguinité de l’ensemble des populations (FIT = 0,848) et du coefficient de consanguinité à l’intérieur des sous-populations (FIS = 0,753) traduisent une déviation des fréquences génotypiques observées chez P. lunatus par rapport aux fréquences attendues dans les conditions de Hardy-Weinberg.
28Marqueurs microsatellites. La diversité génétique totale (HT), la diversité génétique intrapopulation (HS), la diversité génétique interpopulation (DST) et le coefficient de différenciation génique (GST) ont été calculés à partir des fréquences alléliques des loci microsatellites (Tableau 5).
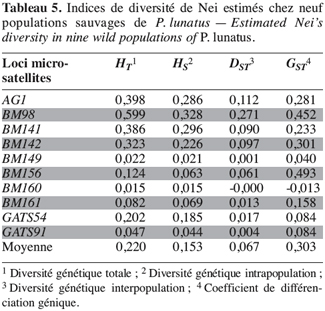
29Les marqueurs microsatellites ont mis en évidence une plus grande diversité génétique totale (HT = 0,220) au sein des populations sauvages de P. lunatus et une plus grande diversité génétique intrapopulation (HS = 0,153) par rapport aux valeurs estimées avec les marqueurs enzymatiques (HT = 0,120 et HS = 0,080). Par contre, la contribution de la diversité génique interpopulation (GST) à la diversité génique totale est identique à celle calculée avec les marqueurs enzymatiques : cette valeur est en moyenne de 30 %.
30L’étude de la variabilité génétique des populations sauvages de P. lunatus avec les marqueurs microsatellites montre que la contribution de la diversité génétique intrapopulation (HS) à la diversité génétique totale (HT) est supérieure à la contribution de la diversité génétique interpopulation (DST = 0,067).
31L’utilisation des marqueurs microsatellites a aussi confirmé l’existence d’une déviation des fréquences génotypiques observées chez P. lunatus par rapport aux fréquences génotypiques attendues dans les conditions de Hardy-Weinberg (FIT = 0,945). Les marqueurs microsatellites montrent que le niveau des croisements non panmictiques à l’intérieur des populations est plus élevé (FIS = 0,916) que celui indiqué par les marqueurs enzymatiques (FIS = 0,753). La différenciation génétique entre les populations FST est égale à 0,346, une valeur légèrement inférieure à celle obtenue avec les marqueurs enzymatiques (FST = 0,386).
4. Discussion et conclusion
4.1. Diversité génétique
32Marqueurs enzymatiques. La forte hétérogénéité des indices de diversité génétique intrapopulation calculés à partir des marqueurs enzymatiques a été mise en évidence chez d’autres espèces autogames (Djè et al., 1999 ; Gao, Hong, 2000). Le pourcentage moyen de loci polymorphes (P) obtenu pour l’ensemble des populations étudiées dans la vallée centrale est de 20,57. Ce pourcentage de loci polymorphes est similaire à la moyenne mentionnée pour les espèces autogames (P = 20,3 %) par Hamrick et Godt (1989). Dans des travaux plus spécifiques portant sur des espèces à autogamie prédominante, signalons une valeur P de 23,90 % ± 8,3 chez Vigna unguiculata (L.) Walp. (Pasquet, 2000) et de 22,6 % ± 6,23 chez Oryza rufipogon Griff. (Gao, Hong, 2000). Toutes ces valeurs observées chez les plantes autogames sont relativement similaires et généralement inférieures à celles observées chez les plantes allogames, comme le maïs où P peut atteindre 96 % (Hamrick, Godt, 1997) ou 94 % (Lu et al., 2002).
33Le faible nombre moyen d’allèles par locus obtenu chez P. lunatus (A = 1,227 ± 0,051) est similaire à celui observé chez des espèces autogames ou à autogamie prédominante : A = 1,320 ± 0,130 chez Oryza rufipogon (Gao, Hong, 2000) ; A = 1,271 ± 0,131 chez Vigna unguiculata (Pasquet, 2000) ; A = 1,566 ± 0,060 chez Vicia tetrasperma (L.) Schreber (Huth, Huth, 2001) et A = 1,250 ± 0,080 chez Sorghum bicolor (L.) Moench. (Djè et al., 1999). A l’intérieur du genre Phaseolus, le nombre moyen d’allèles par locus de P. lunatus observé dans notre étude est similaire à celui rapporté chez une autre espèce autogame, P. vulgaris : A = 1,100 ± 0,100 (Escalante et al., 1994). Par contre, les valeurs de A chez ces deux espèces autogames sont inférieures à celles observées chez une espèce allogame apparentée, comme P. coccineus L., avec A = 2,750 ± 0,210 chez la forme sauvage ssp. formosus et A = 2,650 ± 0,070 chez la forme cultivée ssp. coccineus (Escalante et al., 1994).
34Les valeurs de l’hétérozygotie observée inférieures à celles de l’hétérozygotie attendue, que nous avons obtenues chez toutes les populations analysées de P. lunatus, sont des résultats également rapportés dans une étude réalisée chez une autre plante autogame, le sorgho par Djè et al. (1999). De plus, les valeurs de l’hétérozygotie attendue (He) chez les populations de P. lunatus sont semblables à celles rencontrées chez Sorghum bicolor : He = 0,054 ± 0,022 (Djè et al., 1999), Oryza rufipogon : He = 0,067 ± 0,020 (Gao, Hong, 2000), Vigna unguiculata : He = 0,052 ± 0,017 (Pasquet, 2000) et P. vulgaris : He = 0,041 ± 0,041 (Escalante et al., 1994). Chez l’espèce allogame P. coccineus, on observe par contre, une hétérozygotie attendue élevée qui est respectivement de He = 0,314 ± 0,077 chez la forme sauvage P. coccineus ssp. formosus et de He = 0,296 ± 0,001 chez la forme cultivée P. coccineus ssp. coccineus (Escalante et al., 1994). Les valeurs inférieures observées chez les espèces autogames s’expliquent par la plus faible probabilité de dispersion d’un allèle d’une population à l’autre (Slatkin, 1980). Ce dernier a démontré qu’un nouvel allèle survenant dans une population autogame avait une plus faible probabilité de se répandre dans les autres populations même si cet allèle se caractérisait par une fréquence élevée dans sa population d’origine.
35Marqueurs microsatellites. La grande hétérogénéité des indices de diversité intrapopulation de P. lunatus calculés à partir des marqueurs microsatellites est mise en évidence chez d’autres espèces végétales autogames analysées avec les mêmes marqueurs (Awadalla, Ritland, 1997 ; Djè et al., 1999 ; Djè et al., 2000). Le pourcentage de loci polymorphes (P) chez les populations analysées de P. lunatus est comparable aux valeurs observées chez les espèces autogames Mimulus platycalyx Pennell : P = 67 %, Mimulus laciniatus A. Gray : P = 72 % et Mimulus guttatus Fisch. (ex DC.) var. depauperatus (Gray) A. L. Grant : P = 50 % (Awadalla, Ritland, 1997), ainsi que chez les formes cultivées de Sorghum bicolor : P = 68 % ± 28,3 (Djè et al., 2000).
36Le nombre moyen d’allèles par locus observé chez P. lunatus avec les marqueurs microsatellites reste faible : 1,64 ± 0,384. Il est inférieur à celui obtenu chez Sorghum bicolor : A = 2,43 ± 0,74 (Djè et al., 2000). La différence élevée du nombre moyen d’allèles par locus entre ces deux espèces à système de reproducton semblable s’expliquerait par le fait que le matériel échantillonné par Djè et al. (2000) est composé non seulement de cinq races différentes de Sorghum bicolor, mais aussi de variétés provenant de 18 pays répartis sur trois continents : Afrique, Asie et Amérique.
37La plus petite valeur de l’hétérozygotie observée par rapport à celle de l’hétérozygotie attendue, que nous avons obtenue dans toutes les populations analysées du haricot de Lima, est aussi rapportée chez le sorgho sur base des mêmes marqueurs microsatellites. Les écotypes de Sorghum bicolor originaires du Maroc et collectés in situ possèdent une hétérozygotie observée de 0,272 ± 0,097 et une hétérozygotie attendue de 0,719 ± 0,049 (Djè et al., 1999). Les valeurs de ces indices sont plus faibles chez les accessions de Sorghum bicolor conservées ex situ dans les banques de gènes : Ho = 0,134 ± 0,092 et He = 0,291 ± 0,142 (Djè et al., 2000). Par contre, chez les espèces allogames, les fréquences génotypiques sont plus proches de celles de Hardy-Weinberg (Charlesworth, Wright, 2001).
38Les valeurs généralement plus grandes du nombre moyen d’allèles par locus (A) et de l’hétérozygotie attendue (He) estimées par les marqueurs microsatellites, comparativement aux marqueurs enzymatiques, sont aussi constatées chez d’autres espèces végétales (Desplanque et al., 1999 ; Djè et al., 1999 ; Tanaka et al., 1999 ; Djè et al., 2000 ; Gao, Hong, 2000 ; Pasquet, 2000 ; Sefc et al., 2000 ; Huth, Huth, 2001 ; Hernandez et al., 2002 ; Lu et al., 2002 ; Maguire et al., 2002 ; Shepherd et al., 2002). Les marqueurs microsatellites se caractérisent par un pouvoir discriminant plus élevé.
4.2. Structure génétique
39Marqueurs enzymatiques. Les travaux de Djè et al. (1999) chez Sorghum bicolor, de Pasquet (2000) chez Vigna unguiculata et de Huth et Huth (2001) chez Vicia tetrasperma, trois espèces à autogamie prédominante, ont montré des valeurs d’indices de diversité génétique différentes, tout en aboutissant au même constat que celui obtenu avec notre matériel analysé : la diversité génétique intrapopulation (HS) est supérieure à la diversité génétique interpopulation (DST).
40Le coefficient de différenciation génique (GST) du matériel étudié est élevé et comparable à celui obtenu par Hamrick et Godt (1997) pour les espèces annuelles (GST = 0,357). La diversité génétique intrapopulation de la présente étude est similaire à celle (HS = 0,082) obtenue par Zoro Bi (1999) avec des populations sauvages de P. lunatus originaires de la vallée centrale du Costa Rica. Dans une étude de référence, Hamrick et Godt (1997) montrent que la diversité génétique intrapopulation chez les espèces allogames varie entre 0,103 et 0,266 alors que la diversité génétique intrapopulation des espèces autogames varie entre 0,009 et 0,140 (Hamrick, Godt, 1997).
41Marqueurs microsatellite. La contribution élevée de la diversité génétique intrapopulation (HS) à la diversité génétique totale (HT), observée dans notre matériel, est aussi mise en évidence dans une étude sur la diversité génétique des écotypes de Sorghum bicolor utilisant à la fois les marqueurs enzymatiques et microsatellites (Djè et al., 1999). Compte tenu de cette contribution élevée de la diversité génétique intrapopulation à la diversité génétique totale, les populations sauvages du haricot de Lima peuvent être considérées comme des sources potentielles de réservoir génétique pour la conservation in situ.
5. Conclusions
42L’analyse de plusieurs populations sauvages du haricot de Lima dans la vallée centrale du Costa Rica a montré que les marqueurs microsatellites donnent en général des indices plus élevés de diversité et de structure génétiques, comparativement aux marqueurs enzymatiques. Ceci peut s’expliquer, en partie, par la différence de la richesse allélique entre les deux types de marqueurs en rapport avec la nature du polymorphisme qu’ils révèlent. Les marqueurs enzymatiques révèlent le polymorphisme issu de l’expression des gènes, alors que les marqueurs microsatellites montrent le polymorphisme provenant de la variation des séquences nucléotidiques répétées. Comme le nombre d’allèles de chaque type de marqueur génétique est en relation avec le taux de mutation, la richesse allélique des microsatellites est généralement plus élevée. En effet, le taux de mutation des marqueurs enzymatiques est de l’ordre de 10-6 (Voelker et al., 1980), alors qu’il est de l’ordre de 10-2 à 10-4 chez les marqueurs microsatellites (Tautz, 1989 ; Weber, Wong, 1993). La différence des valeurs d’indices de diversité entre les deux marqueurs peut aussi être liée à l’échantillonnage du matériel végétal analysé. Ainsi, pour les marqueurs microsatellites, toutes les populations étudiées proviennent d’une même région et sont proches les unes des autres ; le nombre d’individus échantillonnés par population est aussi plus élevé. Dans le cas des marqueurs enzymatiques, les populations choisies sont plus dispersées dans la vallée et le nombre d’individus échantillonnés par population est plus restreint. L’étude menée à deux échelles différentes entraînent un biais dans la comparaison des performances des deux marqueurs.
43Néanmoins, l’utilisation des deux marqueurs indique des tendances communes dans l’analyse de la variabilité génétique des populations sauvages de la vallée. Quels que soient les marqueurs utilisés, les populations sauvages du haricot de Lima dans la vallée centrale du Costa Rica se caractérisent par un nombre moyen d’allèles par locus (A) relativement faible, en comparaison de ce qui est observé chez d’autres espèces végétales à autogamie prédominante. Ce nombre peu élevé d’allèles pourrait s’expliquer, d’une part, par l’origine des populations sauvages, toutes restreintes à la vallée centrale du Costa Rica, et, d’autre part, par le fait que seules les amorces microsatellites qui correspondent aux séquences de P. vulgaris conservées chez P. lunatus ont été amplifiées. Or, dans le genre Phaseolus, P. vulgaris et P. lunatus sont phylétiquement très éloignées. La recherche ou la disponibilité d’amorces microsatellites spécifiques à P. lunatus pourraient donner des loci se caractérisant par une richesse allélique plus élevée.
44L’hétérozygotie observée (Ho), tant avec les marqueurs enzymatiques qu’avec les marqueurs microsatellites, est faible et inférieure à l’hétérozygotie attendue (He). La déviation des fréquences génotypiques par rapport aux proportions attendues dans les conditions de l’équilibre de Hardy-Weinberg est manifeste au sein des populations sauvages de P. lunatus originaires de la vallée centrale du Costa Rica. à partir de graines provenant de populations échantillonnées dans la vallée, Masumbuko (1999), Zoro Bi (1999) et Zoro Bi et al. (1999) ont montré que le taux d’allogamie (t) chez cette espèce variait entre 7 et 12,5 % et que l’autogamie observée était le fait de l’autofécondation pour 93 % et de la consanguinité biparentale pour 7 %. Les valeurs élevées du coefficient de consanguinité de l’ensemble des populations (FIT) et du coefficient de consanguinité à l’intérieur des populations (FIS) traduisent aussi une déviation des fréquences génotypiques observées chez le haricot de Lima par rapport aux fréquences attendues dans les conditions de Hardy-Weinberg. Cette déviation est due à des croisements non panmictiques et à une différentiation génétique élevée entre les populations : les valeurs du FST sont similaires avec les deux marqueurs et supérieures à 0,250 (Wright, 1978). Ce résultat reflète une grande divergence génétique entre les populations sauvages de P. lunatus. En considérant l’ensemble des populations sauvages de P. lunatus étudiées par les marqueurs enzymatiques, la valeur du FIT indique que les fréquences génotypiques observées correspondent à celles attendues dans le cas d’une forte autogamie (Charlesworth, Wright, 2001). La diversité génétique au sein des populations sauvages du haricot de Lima de la vallée centrale serait plus le résultat de la présence d’homozygotes possédant des allèles différents que d’hétérozygotes, ce qui est en accord avec le système de reproduction de l’espèce.
45Le système de reproduction est un des principaux facteurs qui influence la fréquence des génotypes dans les populations et qui explique en particulier la consanguinité chez celles-ci (Del Castillo, 1994). Zoro Bi et al. (1999) ont montré que la consanguinité observée chez les populations sauvages du haricot de Lima dans la vallée centrale était essentiellement le fait de l’autofécondation. L’effet majeur de la consanguinité à l’intérieur d’une population est d’accroître le niveau d’homozygotie. Néanmoins, chez des populations végétales maintenues in situ, la réserve en graines du sol peut favoriser la diversité génétique des populations végétales : c’est particulièrement le cas si le système de reproduction de la plante est basé sur l’allogamie et si les graines de la réserve du sol se distinguent par une grande variabilité génétique et maintiennent leur viabilité très longtemps dans le sol (McCue, Holtsford, 1998). Dans notre cas, les populations sauvages du haricot de Lima se caractérisent par une allogamie partielle ; à maturité, les gousses déhiscentes s’ouvrent brusquement et projettent les graines vers le sol, à proximité des plantes-mères, ces graines ne font pas l’objet de prédation par les animaux car elles sont toxiques. Dans un travail antérieur réalisé dans la vallée centrale du Costa Rica, Degreef et al. (2002) ont montré que la réserve en graines du sol chez diverses populations de P. lunatus varie entre trois et cinq graines par m2, pour une durée de vie de celles-ci de trois à quatre ans et pour un taux de germination annuel variant entre 70 et 80 %. La présence de cette banque de graines dans le sol pourrait donc augmenter la taille efficace de la population maintenue in situ et constituer une source d’allèles nouveaux. Une étude comparative de la diversité et de la structure génétiques des graines de cette banque ainsi que des plantes qui ont donné ces graines permettrait de mieux connaître l’influence de la réserve en graines du sol sur la variabilité génétique des populations sauvages de P. lunatus dans la vallée centrale.
46La contribution élevée de la diversité génétique intrapopulation à la diversité génétique totale suggère que les populations individuelles de P. lunatus dans la vallée constituent des unités génétiques valables de conservation in situ. Considérant la structure génétique de l’espèce dans la vallée centrale, un nombre limité de populations ne suffirait pas pour conserver une proportion significative de la diversité présente in situ. Pour évaluer le nombre et la localisation des populations à conserver, il serait utile d’examiner l’existence de groupes de populations plus apparentées et de définir une stratégie d’échantillonnage stratifiée en fonction de ces groupes. Cette objectif nécessiterait la caractérisation des populations de la vallée centrale par divers marqueurs complémentaires (morphologiques, phénologiques, biochimiques et moléculaires) ainsi que la collecte de données d’identification des sites (coordonnées géographiques, climat, sol, végétation environnante). L’analyse de tous ces paramètres permettrait de préciser les combinaisons de facteurs orientant le choix des populations à conserver in situ. Dans l’immédiat, comme les sites naturels des populations sauvages dans cette région sont menacés par la pression démographique, par des modifications brusques de la vocation des terres et par l’intensification de l’agriculture, il est urgent de mener une conservation ex situ couplée avec une conservation in situ. La conservation ex situ concernerait, en priorité, les graines provenant des populations les plus menacées de disparition. Dans chacune des régions climatiques représentatives du Costa Rica, des réserves génétiques qui abritent le maximum de grandes populations devraient être constituées. L’introduction de populations dans les réserves génétiques et leur maintien in situ contribueront à la préservation de l’intégrité génétique et à la capacité d’adaptation du matériel sauvage dans son écosystème.
47Remerciements
48Les auteurs remercient le Pr Oscar Rocha et son équipe de l’Escuela de Biologiá qui ont efficacement collaboré à la collecte des échantillons de P. lunatus dans la vallée centrale du Costa Rica, et la Communauté Française de Belgique pour avoir octroyé une bourse de voyage au Costa Rica à M. Ouédraogo. Des remerciements sont également adressés, d’une part, à la Direction Générale de la Coopération au Développement (DGCD) de la Belgique pour son appui financier au projet de conservation in situ au Costa Rica et, d’autre part, au Centre National de la Recherche Scientifique et Technologique (CNRST) du Burkina Faso pour le financement de la formation de M. Ouédraogo en Belgique.
Bibliographie
Applied Maths. (1997). GelCompar. Comparative analysis of electrophoresis patterns Version 4.2. Available on http://applied-maths.com/home.html.
Awadalla P., Ritland K. (1997). Microsatellite variation and evolution in the Mimulus guttatus species complex with contrasting mating systems. Mol. Biol. Evol. 14, p. 1023–1034.
Bank Hvd., Bank Mvd., Wyk BEv. (2001). A review of use of allozyme electrophoresis in plant systematics. Biochem. Syst. Ecol. 29, p. 469–483.
Baudet JC. (1977). Origine et classification des espèces cultivées du genre Phaseolus. Bull. Soc. R. Bot. Belg. 110, p. 65–76.
Buth DG., Murphy RW. (1999). The use of isozyme characters in systematic studies. Biochem. Syst. Ecol. 27, p. 117–129.
Charlesworth D., Wright SI. (2001). Breeding systems and genome evolution. Curr. Opin. Genet. Dev. 11, p. 685–690.
Crossa J., Hernandez CM., Bretting P., Eberhart SA., Taba S. (1993). Statistical genetic considerations for maintaining germ plasm collections. Theor. Appl. Genet. 86, p. 673–678.
Degreef J., Baudoin JP., Rocha OJ. (1997). Case studies on breeding systems and its consequences for germplasm conservation. 2. Demography of wild Lima populations in the Central Valley of Costa Rica. Genet. Resour. Crop Evol. 44, p. 429–438.
Degreef J., Rocha OJ., Vanderborght T., Baudoin JP. (2002). Soil seed bank and seed dormancy in wild populations of Lima bean (Fabaceae): considerations for in situ and ex situ conservation. Am. J. Bot. 89, p. 1644–1650.
Del Castillo RF. (1994). Factors influencing the genetic structure of Phacelia dubia, a species with a seed bank and large fluctuations in population size. Heredity 72, p. 446–458.
Desplanque B., Boudry P., Broomberg K., Saumitou-Laprade P., Cuguen J., Van Dijk H. (1999). Genetic diversity and gene flow between wild, cultivated and weedy forms of Beta vulgaris L. (Chenopodiaceae), assessed by RFLP and microsatellite markers. Theor. Appl. Genet. 98, p. 1194–1201.
Djè Y., Forcioli D., Ater M., Lefèbvre C., Vekemans X. (1999). Assessing population genetic structure of sorghum landraces from north-western Morocco using allozyme and microsatellite markers. Theor. Appl. Genet. 99, p. 157–163.
Djè Y., Heuertz M., Lefèbvre C., Vekemans X. (2000). Assessment of genetic diversity within and among germplasm accessions in cultivated sorghum using microsatellites markers. Theor. Appl. Genet. 100, p. 918–925.
Escalante AM., Coello G., Eguiarte LE., Pinero D. (1994). Genetic structure and mating systems in wild and cultivated populations of Phaseolus coccineus and P. vulgaris (Fabaceae). Am. J. Bot. 81, p. 1096–1103.
Fahima T., Röder MS., Wendehake K., Kirzhner VM., Nevo E. (2002). Microsatellite polymorphism in natural populations of wild emmer wheat, Triticum dicoccoides, in Israel. Theor. Appl. Genet. 104, p. 17–29.
Ford R., Le Roux K., Itman C., Brouwer JB., Taylor PWJ. (2002). Diversity analysis and genotyping in Pisum with sequence tagged microsatellite site (STMS) primers. Euphytica 124, p. 397–405.
Gao Lz., Hong SGDy. (2000). Allozyme variation and population genetic structure of common wild rice Oryza rufipogon Griff. in China. Theor. Appl. Genet. 101, p. 494–502.
Goudet J. (1995). Fstat (ver 1.2): a computer program to calculate F-statistics. J. Hered. 86, p. 485–486.
Hamrick Jl., Godt JW. (1989). Allozyme diversity in plant species. In Brown AHD., Cleg MT., Kalher AL., Weir BS. (Eds). Plant population genetics, breeding and genetic resources. Sunderland, Massachussets, USA: Sinauer Associates, p. 43–63.
Hamrick Jl., Godt JW. (1997). Allozyme diversity in cultivated crops. Crop Sci. 37, p. 26–30.
Hardy OJ., Dubois S., Zoro Bi I., Baudoin JP. (1997). Gene dispersal and its consequences on the genetic structure of wild populations of Lima bean (Phaseolus lunatus L.) au Costa Rica. Plant Genet. Resour. Newsl 109, p. 1–6.
Hernandez P., Laurie DA., Martin A., Snape JW. (2002). Utility of barley and wheat simple sequence repeat (SSR) markers for genetic analysis of Hordeum chilense and tritordeum. Theor. Appl. Genet. 104, p. 735–739.
Huth MK., Huth HW. (2001). Genetic diversity and population structure of wild lentil tare. Crop Sci. 41, p. 1940–1946.
Lodieu J. (1997). Développement d'une stratégie de conservation in situ du Phaseolus lunatus L. dans la Vallée Centrale du Costa Rica : intégration du système d'information géographique. Travail de fin d’études. Gembloux, Belgique : Faculté Universitaire des Sciences agronomiques.
Lu HL., Li JS., Liu JL., Bernado R. (2002). Allozyme polymorphisms of maize populations from southwestern China. Theor. Appl. Genet. 104, p. 119–126.
Maguire TL., Peakall R., Saenger P. (2002). Comparative analysis of genetic diversity in the mangrove species Avicennia marina (Forsk.) Vierh. (Avicenniaceae) detected by AFLPs and SSRs. Theor. Appl. Genet. 104, p. 388–398.
Maquet A. (1995). Etude de la diversité génétique de la légumineuse alimentaire Phaseolus lunatus L. par l'analyse des caractères morphophysiologiques et de marqueurs protéiques. Doctorat. Gembloux, Belgique : Faculté universitaire des Sciences agronomiques.
Maquet A., Zoro Bi I., Delvaux M., Wathelet B., Baudoin JP. (1997). Genetic structure of a Lima bean base collection using allozyme markers. Theor. Appl. Genet. 95, p. 980–991.
Masumbuko B. (1999). Etude du flux génique et de la distribution spatiale des gènes chez les populations sauvages du haricot de Lima, Phaseolus lunatus L., originaires de la vallée centrale du Costa Rica. Travail de fin d’études. Gembloux, Belgique : Faculté Universitaire des Sciences agronomiques.
McCue KA., Holtsford TP. (1998). Seed bank influences on genetic diversity in the rare annual Clarkia springvillensis (Onagraceae). Am. J. Bot. 85, p. 30–36.
Meurrens F., Degreef J., Rocha OJ., Baudoin JP. (2001). Demographic study in microconservation sites to maintain in situ wild Lima beans (Phaseolus lunatus L.) in the Central Valley of Costa Rica. Plant Genet. Resour. Newsl. 128, p. 45–50.
Murphy RW., Sites JW., Buth JR., Buth DG., Haufler CH. (1990). Proteins I: Isozyme electrophoresis. In Hillis DM., Moritz C. (Eds). Molecular systematics. Sunderland, Massachusetts USA: Sinauer Associates, p. 45–126.
Ouédraogo M. (2003). Etude de la variabilité génétique et du flux de gènes chez des populations sauvages de Phaseolus lunatus L. dans la vallée centrale du Costa Rica à l’aide des marqueurs enzymatiques et microsatellites. Doctorat. Gembloux, Belgique : Faculté Universitaire des Sciences Agronomiques de Gembloux.
Pasquet RS. (2000). Allozyme diversity of cultivated cowpea Vigna unguiculata (L.) Walp. Theor. Appl. Genet. 101, p. 211–219.
Rocha OJ., Macaya G., Baudoin JP. (1997). Causes of local extinction and recolonization, determined by 3 years of monitoring wild populations of Phaseolus lunatus L. in the Central Valley of Costa Rica. Plant Genet. Resour. Newsl. 112, p. 44–48.
Sefc KM., Lopes MS., Lefort F., Botta R., Roubelakis-Angelakis KA., Ibanez J., Pejié I., Wagner HW., Glossel J., Steinkellner H. (2000). Microsatellite variability in grapevine cultivars from different European regions and evaluation of assignment testing to assess the geographic origin of cultivars. Theor. Appl. Genet. 100, p. 498–505.
Shepherd M., Cross M., Maguire TL., Dieters MJ., Williams CG., Henry RJ. (2002). Transpecific microsatellites for hard pines. Theor. Appl. Genet. 104, p. 819–827.
Slatkin M. (1980). The distribution of mutant alleles in a subdivided population. Genetica 95, p. 503–524.
Stachel M., Lelley T., Grausgruber H., Vollmann J. (2000). Application of microsatellites in wheat (Triticum aestivum L.) for studying genetic differentiation caused by selection for adaptation and use. Theor. Appl. Genet. 100, p. 242–248.
Tanaka K., Tsumura Y., Nakamura T. (1999). Development and polymorphism of microsatellite markers for Fagus crenata and the closely related species, F. japonica. Theor. Appl. Genet. 99, p. 11–15.
Tautz D. (1989). Hypervariability of simple sequence repeats as a general source for polymorphic markers. Nucleic Acids Resour. 17, p. 6463–6471.
Voelker RA., Schaffer HE., Mukai T. (1980). Spontaneous allozyme mutations in Drosophila melanogaster: rate of occurrence and nature of the mutants. Genetics 94, p. 961–968.
Weber JL., Wong C. (1993). Mutation of human short tandem repeats. Hum. Mol. Genet. 2, p. 1123–1128.
Wright S. (1978). Evolution and the genetics of populations. Vol 4. Variability within and among natural populations. Chicago: University of Chicago Press.
Yeh FC., Boyle TJB., (1997). Population genetic analysis of co-dominant and dominant markers and quantitative traits. Belg. J. Bot. 129, p. 157.
Zoro Bi I. (1999). Variabilité génétique des populations sauvages de Phaseolus lunatus L. dans la vallée centrale du Costa Rica et ses implications dans la mise au point d'une stratégie de conservation in situ. Doctorat. Gembloux, Belgique : Faculté universitaire des Sciences agronomiques de Gembloux.
Zoro Bi I., Maquet A., Wathelet B., Baudoin JP. (1997). Genetic control of alcohol dehydrogenase, malate dehydrogenase, and phosphoglucomutase isozymes in lima bean (Phaseolus lunatus L.). Plant Breed. 116, p. 181–185.
Zoro Bi I., Maquet A., Degreef J., Wathelet B., Baudoin JP. (1998). Sample size for collecting seeds in germplasm conservation: the case of the Lima bean (Phaseolus lunatus L.). Theor. Appl. Genet. 97, p. 187–194.
Zoro Bi I., Maquet A., Wathelet B., Baudoin JP. (1999). Genetic control of isozymes in the primary gene pool Phaseolus lunatus L. Biotechnol. Agron. Soc. Environ. 3, p. 10–27.

